1.4. 収録データへのアクセス
https://jmorp.megabank.tohoku.ac.jp へアクセスすると、jMorp のトップページが現れます。
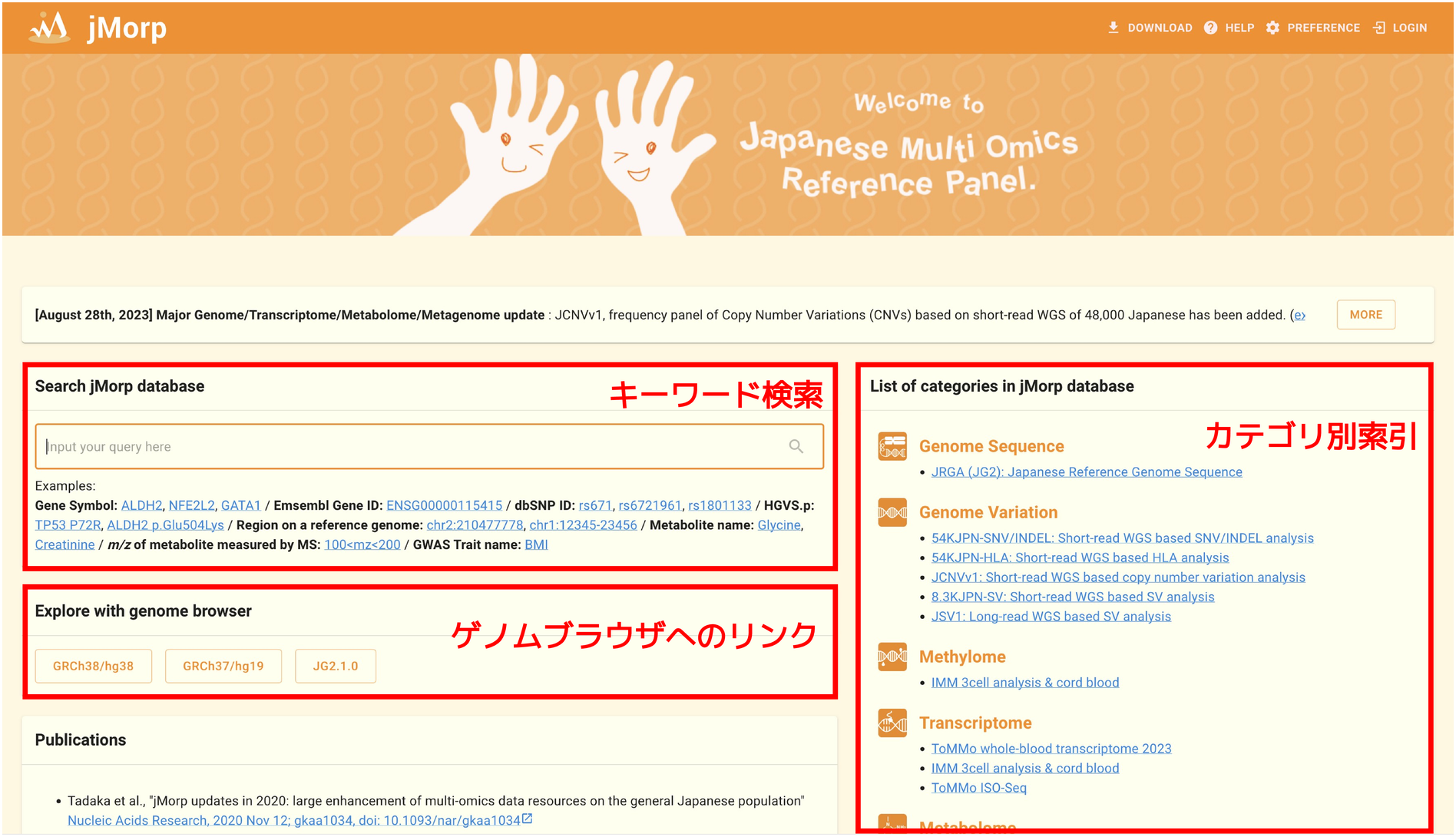
トップページは大きく3ブロック(キーワード検索・ゲノムブラウザ・カテゴリ別索引)に分かれています。
1.4.1. キーワードによる検索
左上に位置する キーワード検索 パネルを利用すると、jMorpに収録されている全データに対してキーワード検索を実行することができます。 キーワードとしては以下のようなものが利用可能です
注釈
下の検索キーワード例をクリックすると、実際の検索結果ページにジャンプします。 どんなページが現れるのか是非試してみて下さい。
カテゴリ |
クエリ例 |
|---|---|
遺伝子シンボル |
|
Ensembl Gene ID |
|
UniProt ID |
|
PDB ID |
|
dbSNP ID (rsID) |
|
遺伝子シンボル + HGVS.p, RefSeq ID + HGVS.p |
TP53 P72R / ALDH2 p.Glu504Lys / NP_000537 P72R / NP_000681 p.Glu504Lys |
ゲノム座標・領域 |
|
代謝物名 |
|
MSで測定された代謝物のm/z, retention time |
|
GWASのトレイト名 |
例えば Creatinine で検索を実行すると、本書執筆時点では以下のような結果が得られます。

検索結果ページの上部はメタボロームデータセットにおける検索結果です。 メタボロームデータセットでは、 Creatinine を NMR ・ LC-MS ・ GC-MS など数種類の手法で測定しており、 それらがヒットしています。
検索結果ページの下部はGWASデータセットにおける検索結果です。 このパネルにあるリンクを辿ると、jMorp に登録されているGWAS解析結果の中からGWASのトレイトとして Creatinine が使われている解析の結果を見ることができます。 この例のように、jMorpのトップページにあるキーワード検索パネルを利用することで、 jMorpに収録されているデータに対し横断して検索を実行することが可能です。
注釈
Creatinine は複数のエントリがヒットするため、前述のような検索結果ページが表示されますが、 入力されたキーワードがjMorpの単一のエントリにしかヒットしなかった場合は検索結果ページが表示されず、 直接ヒットしたエントリのページへジャンプします。
1.4.2. ゲノムブラウザによるデータ表示
jMorpトップページの左下のリンクを辿ることで、ゲノム関連データをゲノムブラウザ形式で閲覧することができます。 本書執筆時点では GRCh37/hg19 ・ GRCh38/hg38 ・ JG2.1.0 3種類のゲノムブラウザを用意しています。 jMorpに収録されているゲノム関連データは解析に利用されている参照ゲノムが異なり、ゲノムブラウザごとに閲覧できるデータが異なります。
- GRCh37/hg19
- Genome Variation
3.5KJPNv2: 短鎖全ゲノム解析 3,500 検体による SNV/INDEL 頻度パネル
4.7KJPN: 短鎖全ゲノム解析 4,700 検体による SNV/INDEL 頻度パネル
8.3KJPN: 短鎖全ゲノム解析 8,300 検体による SNV/INDEL 頻度パネル
8.3KJPN-SV: 短鎖全ゲノム解析 8,3 検体による 構造多型 (Structual Variation; SV) 頻度パネル
- Genome (others)
Genome Accessibility: 短鎖全ゲノム解析の平均深度データ
- GRCh38/hg38
- Genome Variation
14KJPN: 短鎖全ゲノム解析 14,000 検体による SNV/INDEL 頻度パネル
38KJPN: 短鎖全ゲノム解析 38,000 検体による SNV/INDEL 頻度パネル
54KJPN: 短鎖全ゲノム解析 54,000 検体による SNV/INDEL 頻度パネル
JCNVv1: 短鎖全ゲノム解析 48,000 検体による CNV 頻度パネル
JSV1: 長鎖全ゲノム解析 222 検体による SV 頻度パネル
- Genome (others)
Genome Accessibility: 短鎖全ゲノム解析の平均深度データ
- JG2.1.0
- Genome Sequence
JG2.1.0: JG2.1.0 日本人基準配列
- Genome Variation
38KJPN: 短鎖WGS 38,000検体によるSNV/INDEL頻度パネル (GRCh38版からのLiftOver)
54KJPN: 短鎖WGS 54,000検体によるSNV/INDEL頻度パネル (GRCh54版からのLiftOver)
ゲノムブラウザの利用方法については ゲノムブラウザ を参照して下さい。
1.4.3. カテゴリ別索引
jMorpトップページの右側には、jMorpに収録されているデータセットの一覧が並んでいます。 データセット名をクリックすると、そのデータセットに収録されているエントリの一覧を見ることができます。 例えば、 Metabolome 2023 をクリックすると下のようなテーブルが表示され、メタボロームデータセットに含まれる代謝物の一覧を見ることができます。
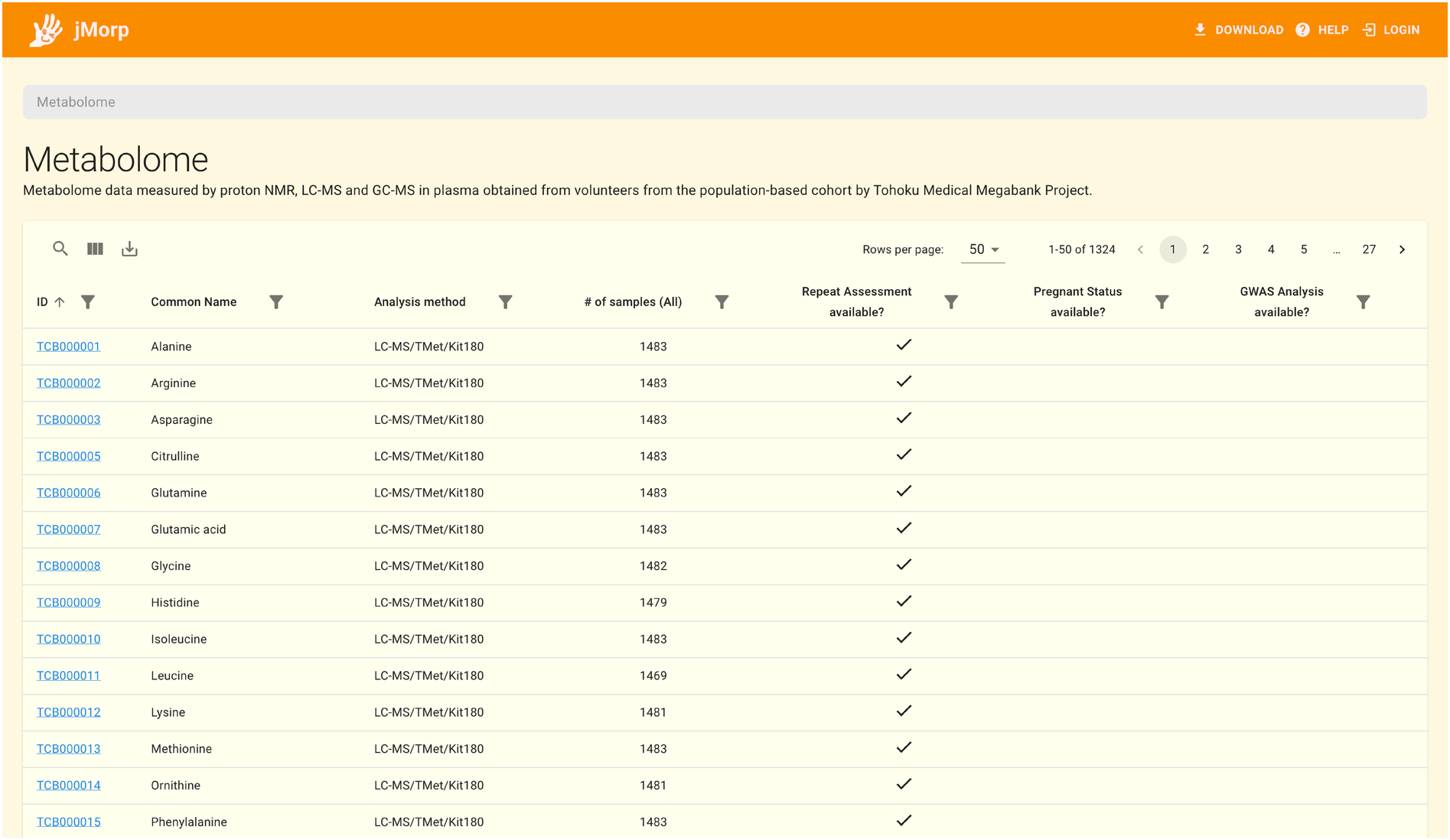
更に代謝物IDをクリックすることで、その代謝物の詳細情報を閲覧することができます。
「jMorp にはどんなデータが入っているのかざっと眺めたい」というときにはカテゴリ別の索引を辿るのが良いでしょう。
注釈
Genome Variation (SNV/INDEL) など、エントリ数が多いカテゴリについては一覧は表示されません。 キーワードによる検索 または ゲノムブラウザによるデータ表示 をご利用下さい。