2.7. ゲノムブラウザ
jMorpにはゲノムブラウザが搭載されており、全ゲノムリファレンスパネルやジャポニカアレイのマーカーの位置、 GWASの結果などをゲノム上の位置関係を確認しながら閲覧することができます。このゲノムブラウザは JBrowse2 を 利用しています。
2.7.1. ゲノムブラウザを開く
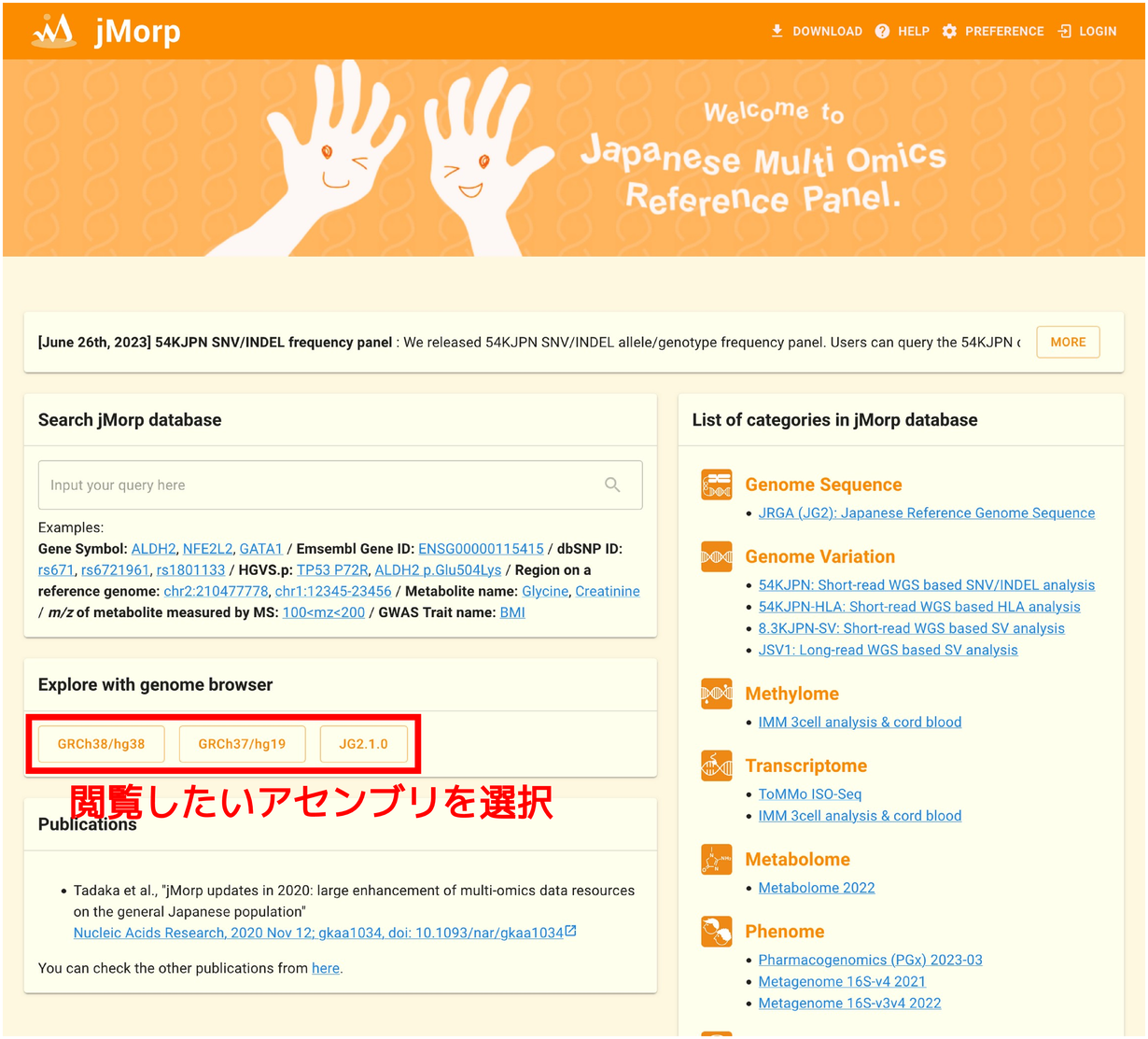
ゲノムブラウザはトップページの「Explore with genome browser」パネルから表示することができます。 「GRCh38/hg38」、「GRCh37/hg19」および「JG2.1.0」の3種類のアセンブリから利用したいものを選ぶことでゲノムブラウザが開きます。
それぞれのアセンブリで提供している情報が異なります。以下に例を示します。
アセンブリ |
提供している情報 |
|---|---|
GRCh38/hg38 |
|
GRCh37/hg19 |
|
JG2.1.0 |
|
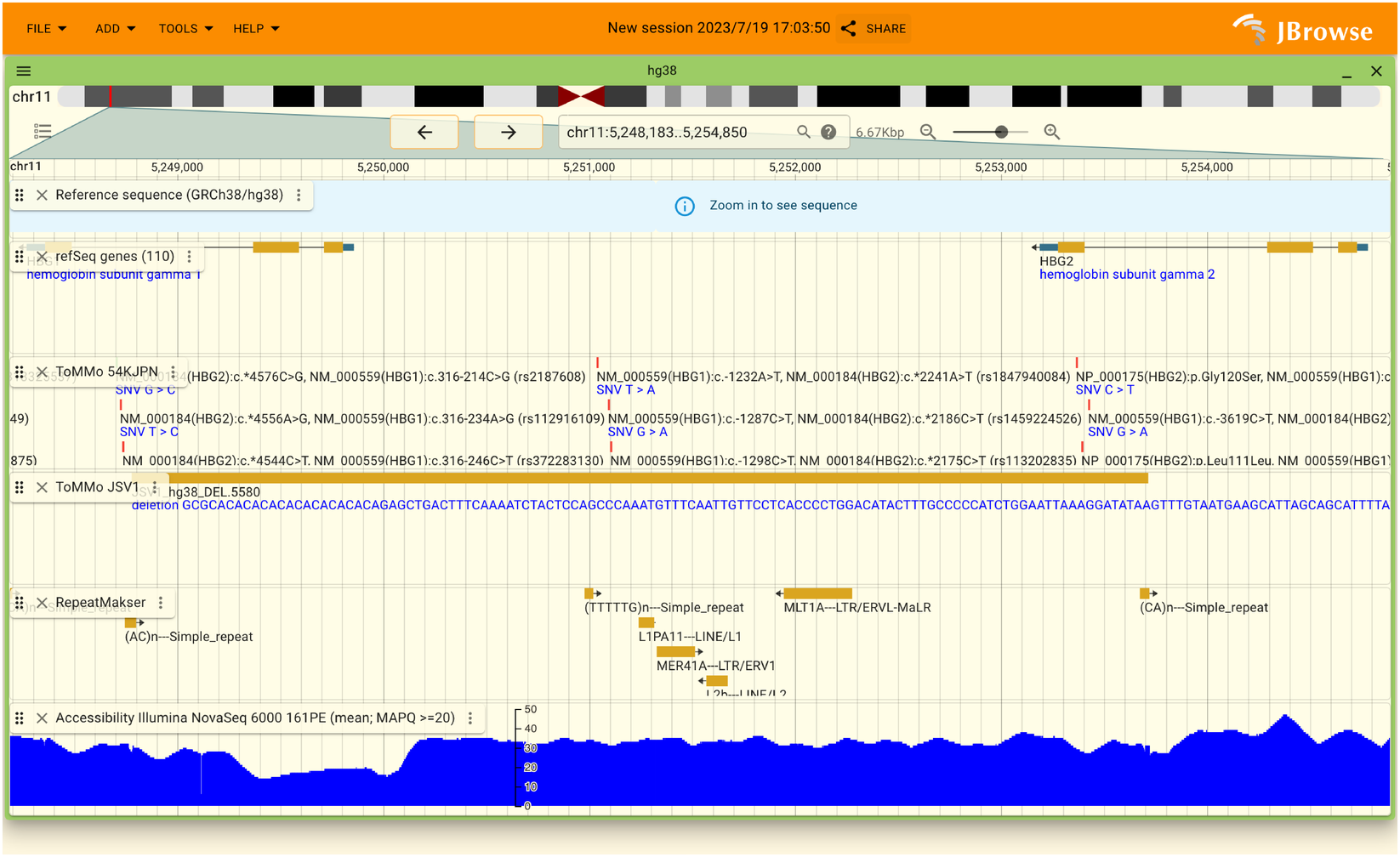
ゲノムブラウザが正常に読み込まれると以下のような表示になります。
2.7.2. トラックの一覧
ゲノムブラウザを開いた直後は遺伝子、全ゲノムリファレンスパネル、長鎖リード版構造多型リファレンスパネル、 Repeat Masker、Accessibilityのみが表示された状態となっています。 jMorpのゲノムブラウザではその他にも様々な情報を表示できます。 それぞれの情報が表示された行はトラックと呼びます。
表示できるトラックの一覧を確認するには以下の画像の赤枠で囲った領域をクリックします。
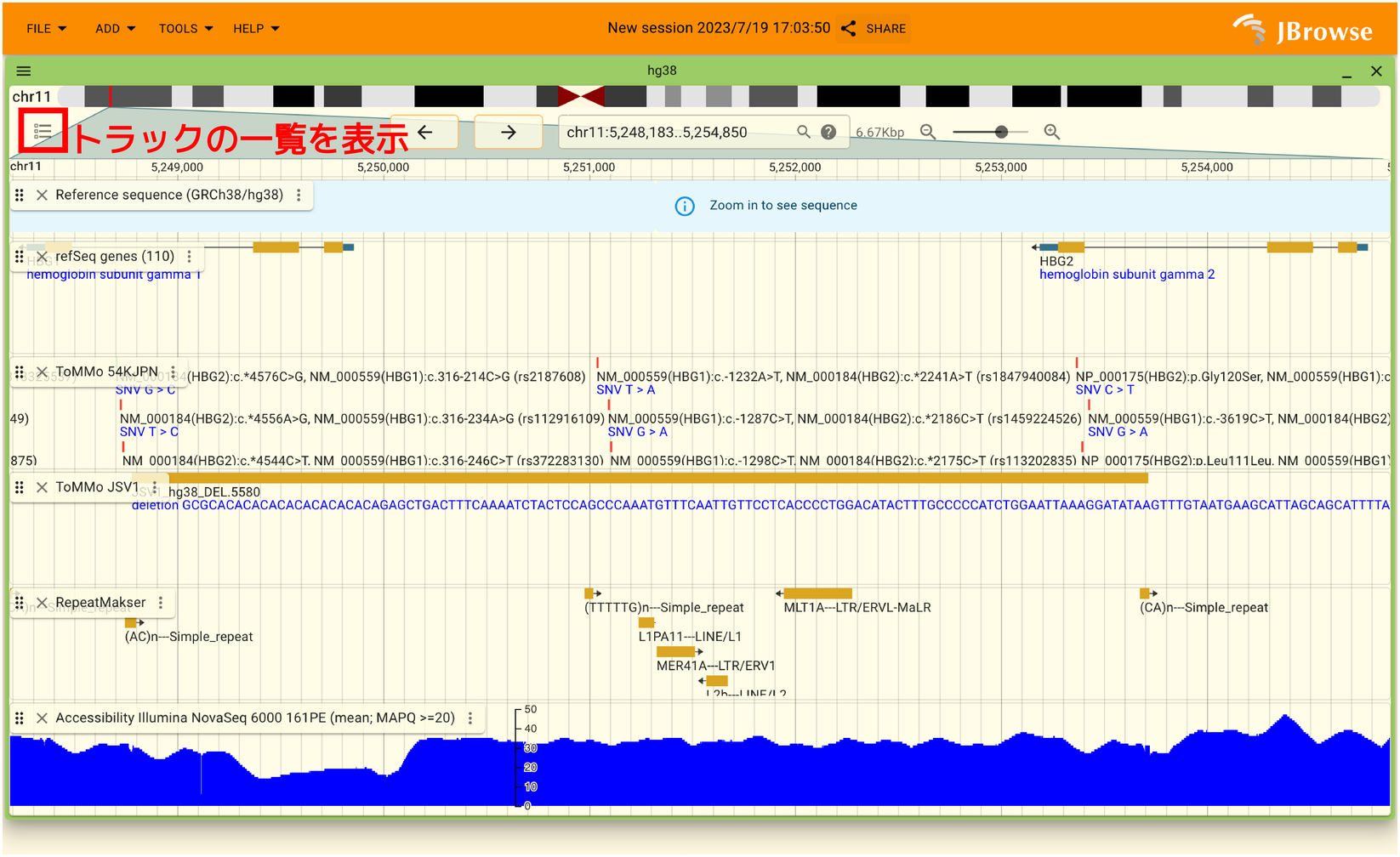
このようにすると以下のようにトラックの一覧が表示されます。
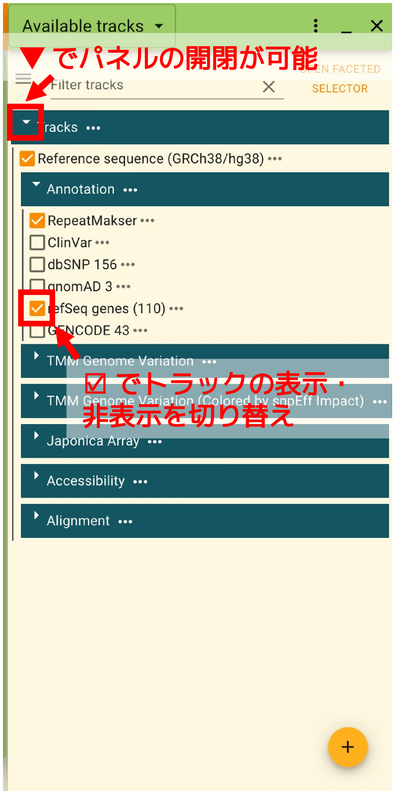
この画面ではトラックの選択ができます。トラックは各カテゴリごとに分類されています。 分類は以下のとおりです。
カテゴリ名 |
内容 |
アセンブリ |
|---|---|---|
Annotation |
|
GRCh38/hg38, GRCh37/hg19, JG2.1.0 |
TMM Genome Variation |
|
GRCh38/hg38, GRCh37/hg19, JG2.1.0 |
TMM Genome Variation (Colored by snpEff Impact) |
|
GRCh38/hg38, GRCh37/hg19 |
Japonica Array |
|
GRCh38/hg38, GRCh37/hg19 |
Accessibility |
|
GRCh38/hg38, GRCh37/hg19 |
Variant co-occurrence |
|
GRCh38/hg38, GRCh37/hg19 |
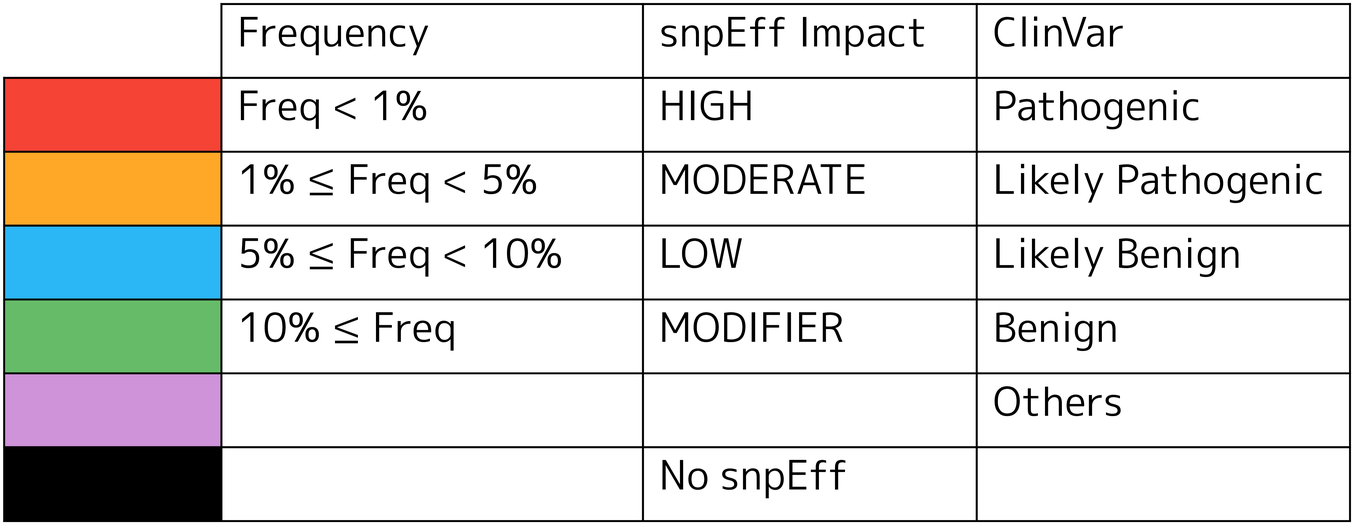
2.7.2.1. 各Variantの色の意味
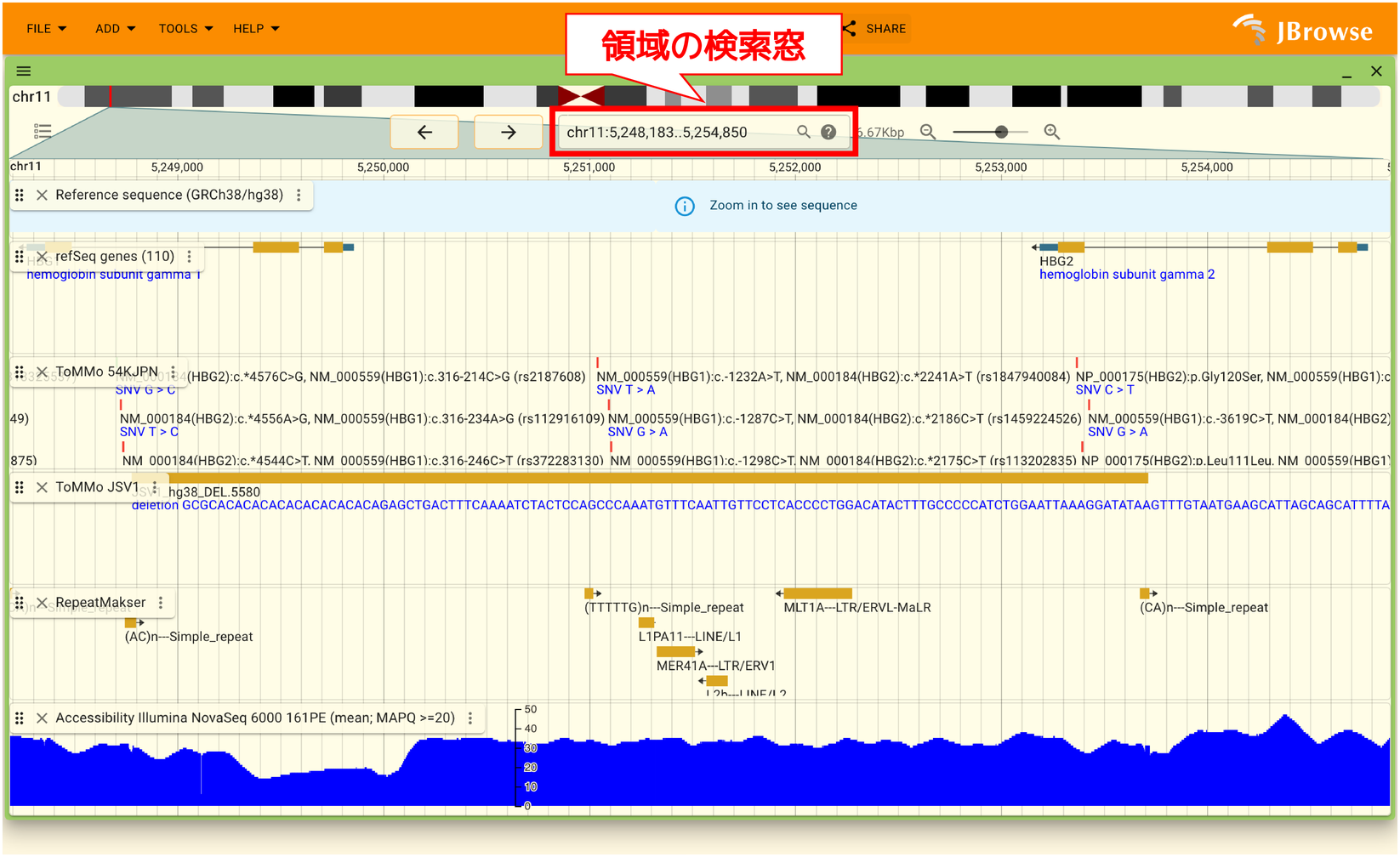
2.7.3. 領域の検索
jMorpのゲノムブラウザでは遺伝子名やdbSNPのrs番号、HGVSによるアミノ酸変化で検索が可能です。 検索は以下の画像のように染色体名やポジションが表示されているボックスから検索ができます。
検索可能な項目と例は以下の通りです。
検索項目 |
検索例 |
注意事項 |
|---|---|---|
遺伝子名 |
|
NRF2などsynonymでは検索できません |
dbSNP rs番号 |
rs671 |
|
HGVSによるアミノ酸変化 |
|
|
もし、複数の領域がヒットした場合には以下のような選択画面となります。
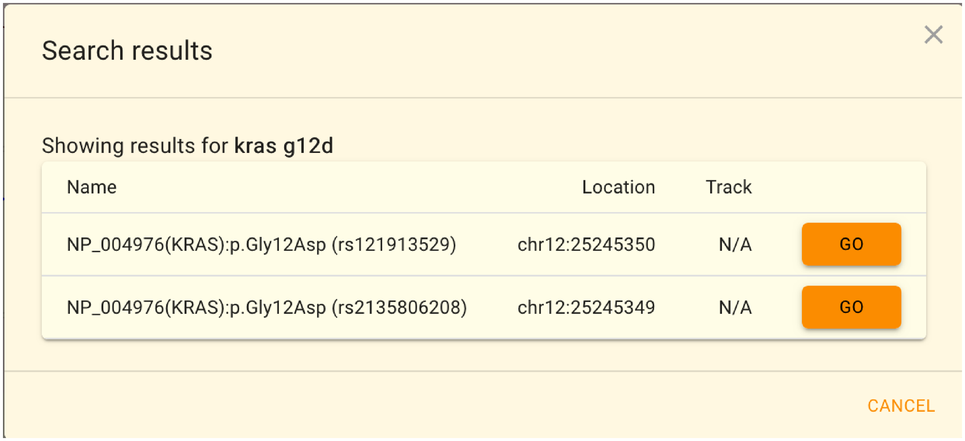
dbSNPのrs番号とHGVSによるアミノ酸変化での検索はGRCh37/hg19もしくはGRCh38/hg38でのみ利用可能であり、 jMorpのJBrowse2特有の機能となっています。JGシリーズでは遺伝子名の検索のみ利用可能です。
2.7.4. 自分のデータを重ねて表示する
JBrowse2では手元にある自分のデータを重ねて表示できます。この時に手元のデータがサーバーにアップロードされることはありませんし、 どのようなデータが表示されているのか東北メディカル・メガバンク機構に通知されることもありません。
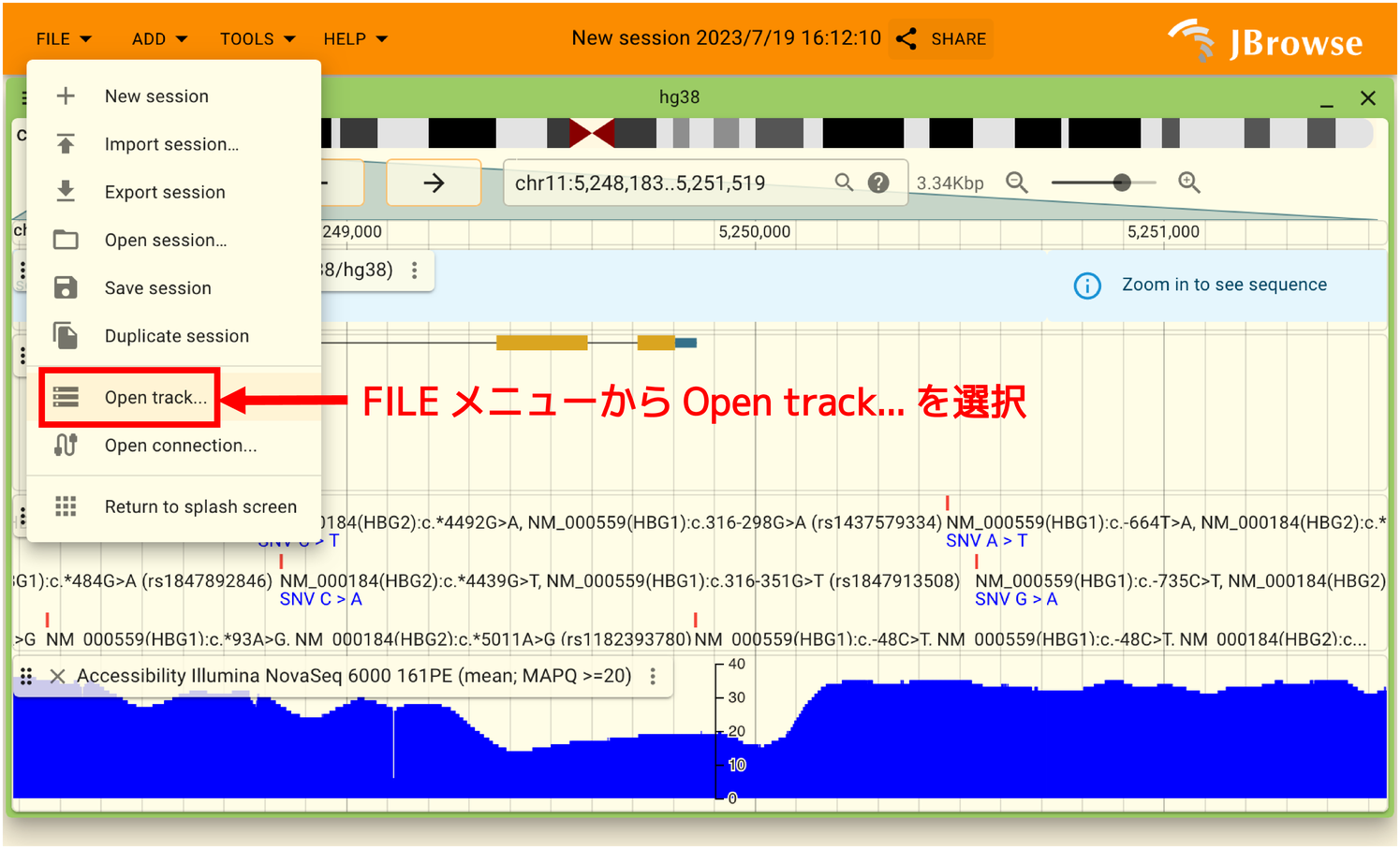
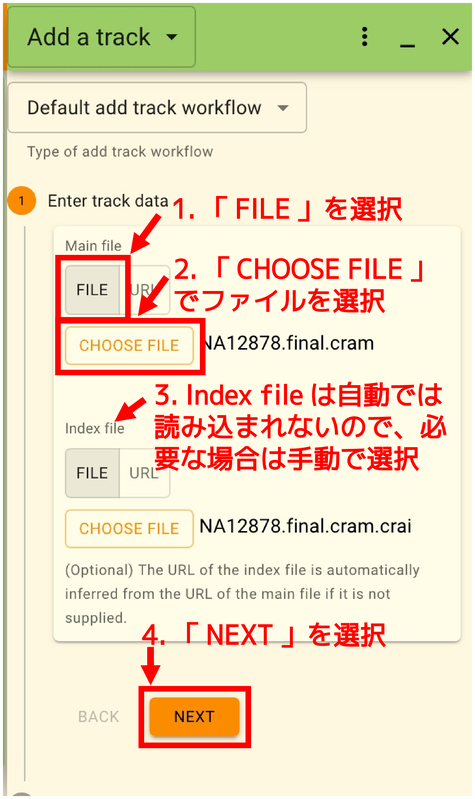
自分のデータを表示させるためには「FILE」メニューから「Open track...」を選択します。
次に右側に表示された「Add a track」パネルでURLを入力するかファイルを選択します。 インデックスファイルが必要なフォーマットの場合、IGVのように自動的に読み込まれることはないので、手動で選択する必要があります。 完了したら「NEXT」をクリックします。
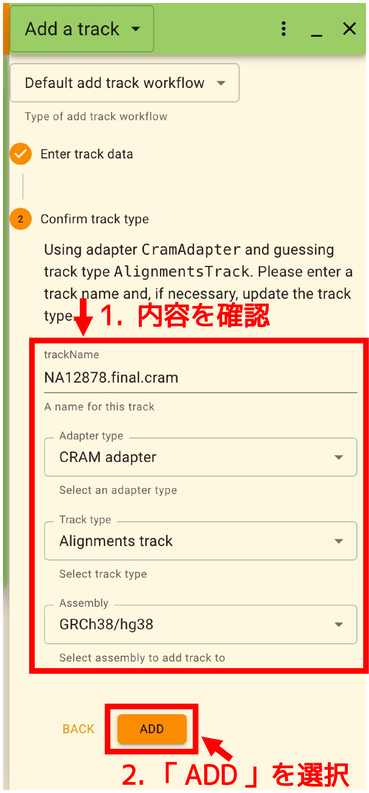
ファイルの内容から必要な設定が自動的に表示されます。問題がなければ「ADD」をクリックします。
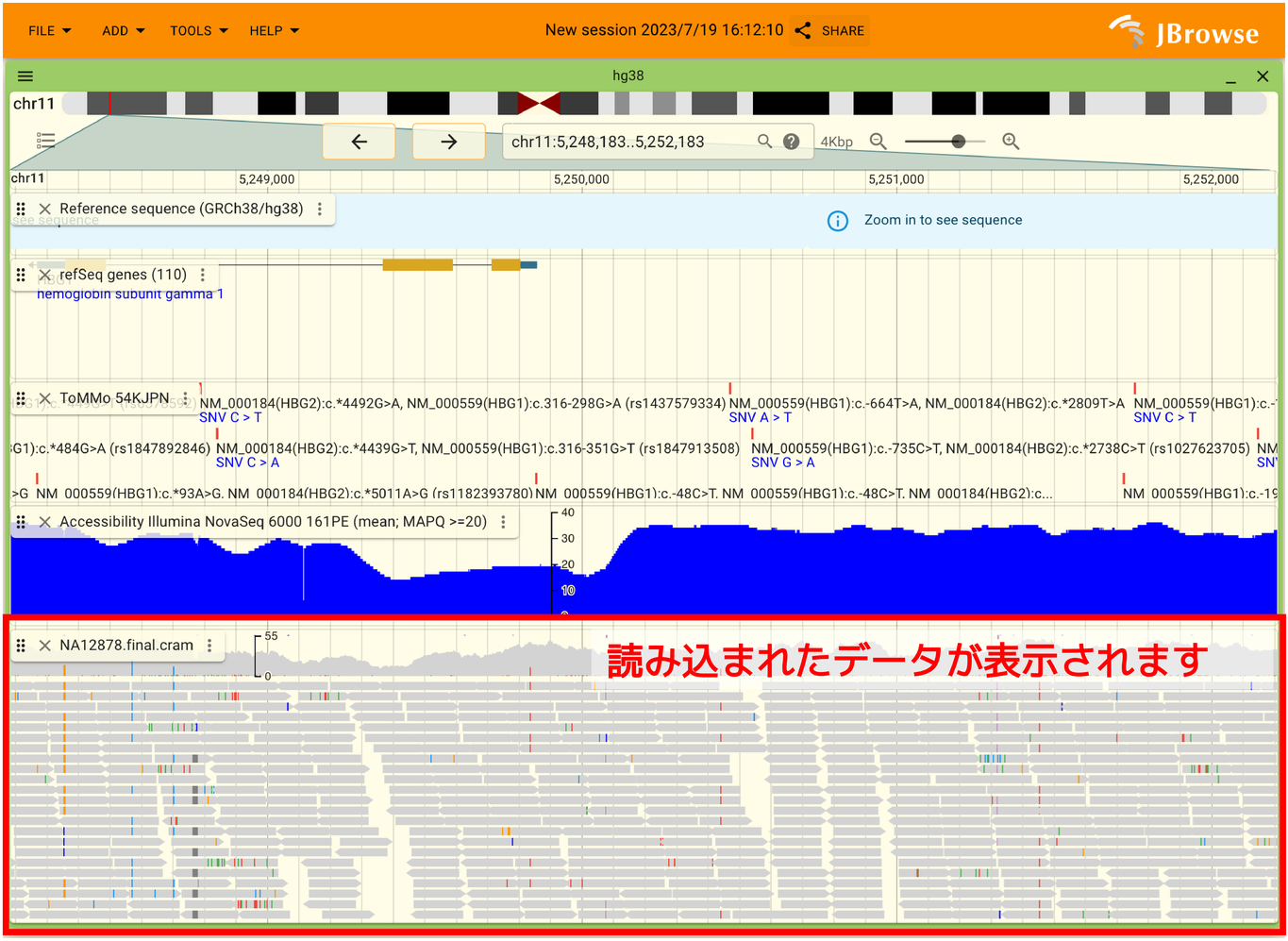
以上の操作を行うと以下のように自分の手元のデータを表示することができます。この例ではCRAMファイルを表示しましたが、BEDファイルやGFFファイルも表示可能です。
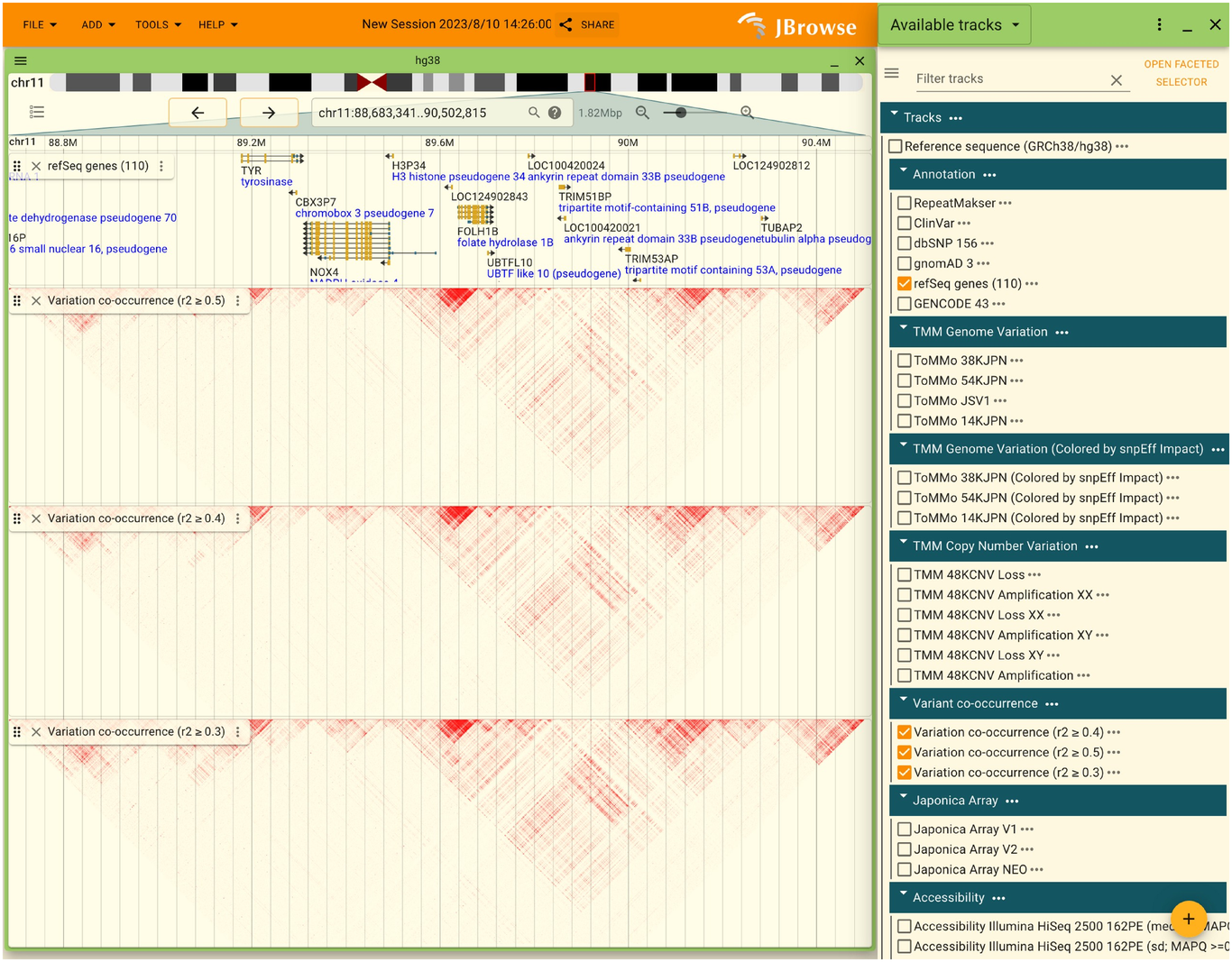
2.7.5. Variant co-occurrenceの表示方法
Variant co-occurrenceは十分に広い領域を表示した時にのみ正しく表示可能です。個々のSNVやINDELが表示できるほど拡大している場合には縮小して表示してください。正しく表示できた場合には以下のような表示になります。
2.7.6. 表示内容を他の人と共有する
JBrowse2では現在表示されている内容を他の人と共有することができます。この時には JBrowse の開発元に現在の表示内容を暗号化したデータが送信されます。 共有するためには以下のように上のヘッダーにある「SHARE」ボタンをクリックします。
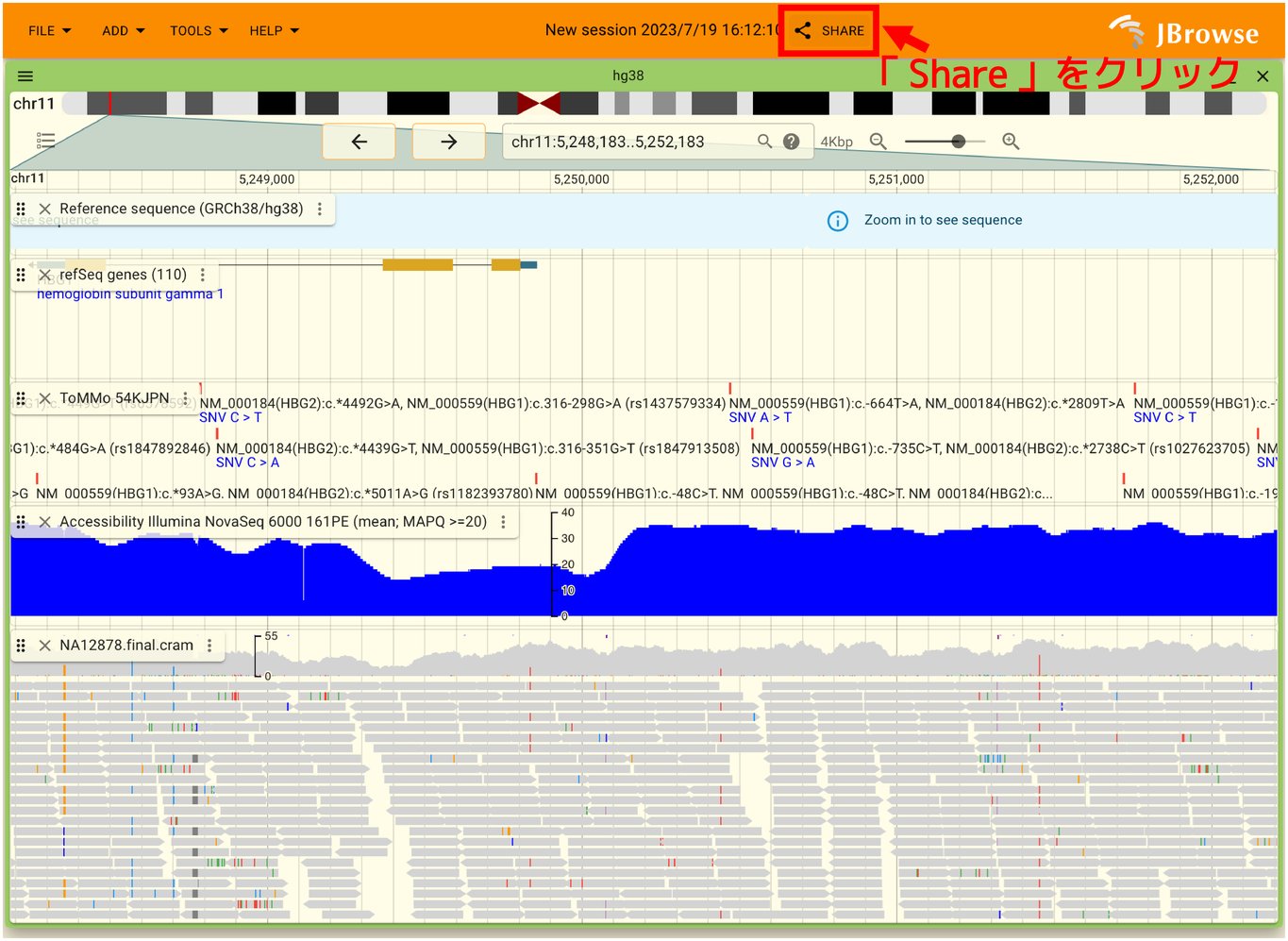
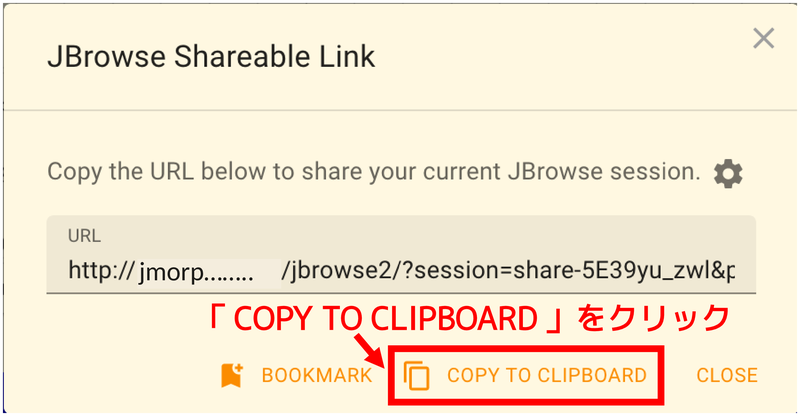
ダイアログが表示されて少し待つと以下のように共有用のURLが表示されます。「COPY TO CLIPBOARD」をクリックするとURLをコピーできますので、このURLを共有してください。
2.7.7. 複数のリファレンスゲノムを比較する
JBrowse2では複数のリファレンスゲノムを比較することができます。
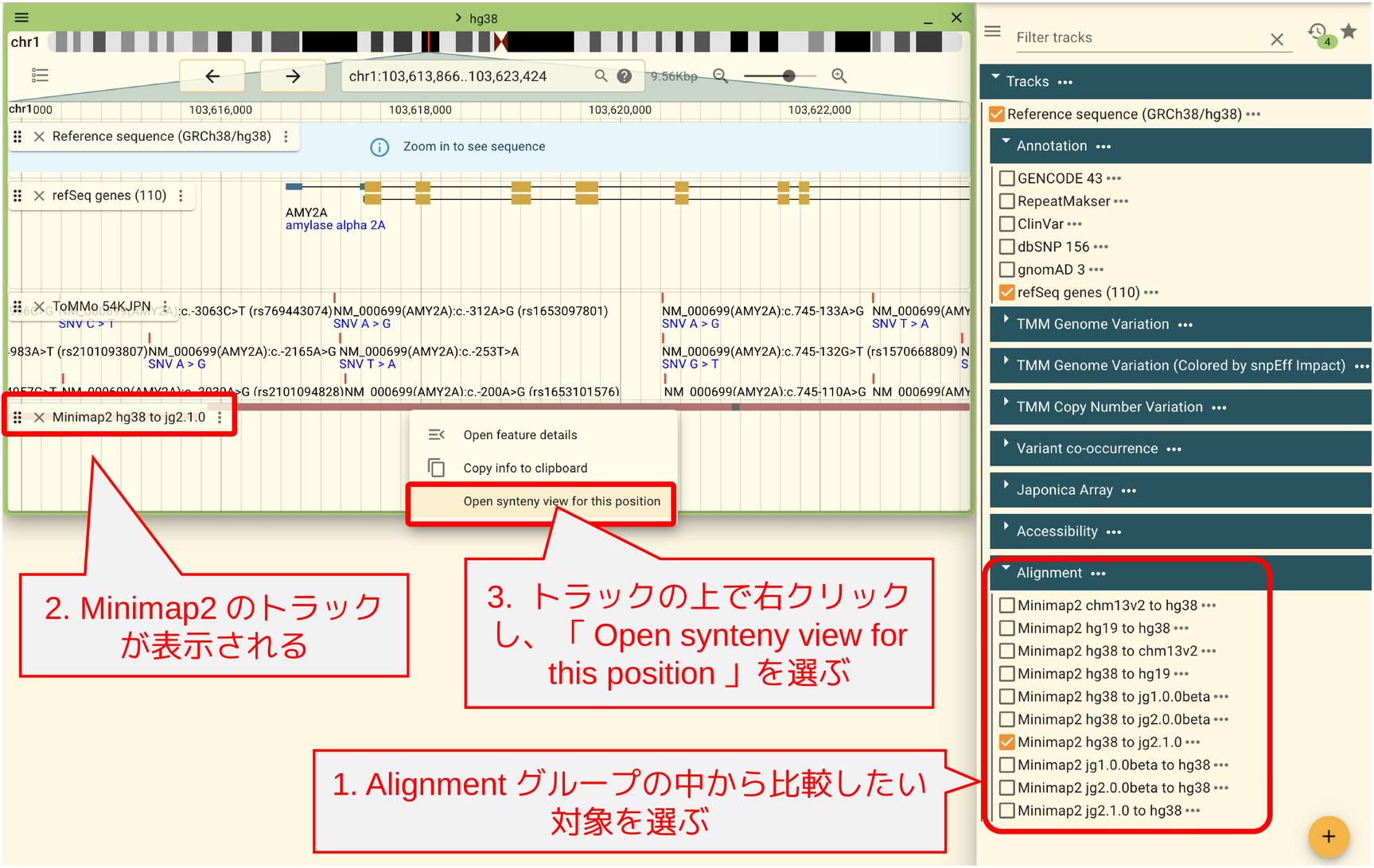
まず、トラックの一覧を開き、Aligmentの中にある「Minimap2 XX to YY」のトラックを開きます。 この時にXXは現在開いているリファレンスゲノム、YYは比較対象のリファレンスゲノムとします。 例えばhg38のリファレンスゲノムを開いていて、JG2.1.0と比較したい時には「Minimap2 hg38 to jg2.1.0」を選択します。 Minimpa2を使ったアライメントはXX to YYとYY to XXの2つのパターンで行なっているため、 同じリファレンスゲノム同士でも両方存在し、若干結果も異なります。ただし、LiftOver用の chainファイルはXX to YYの方で用意していますので、通常はXX to YYを利用してください。
Minimap2のトラックが表示されると以下のような画面になりますので、アライメントが取れている 領域で右クリックし、メニューから「Open synteny view for this position」を選択します。
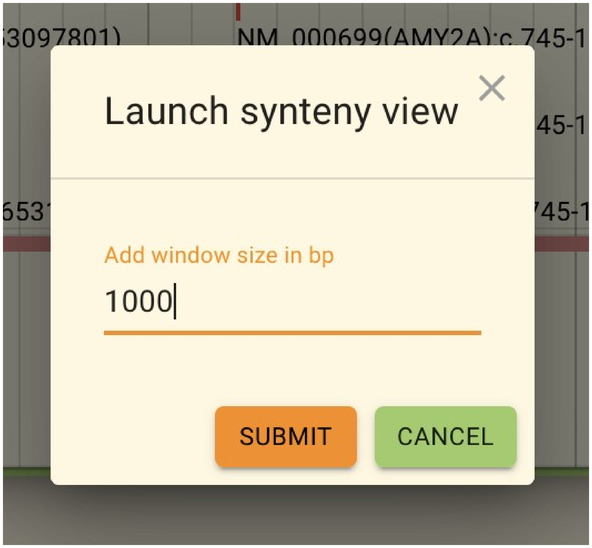
以下の画面では現在閲覧している領域からどれだけ広げるかを設定します。 すでに広い範囲を見ている場合には0としても問題ありません。 数値を設定したら「Submit」をクリックします。
ここまで操作すると以下のような画面になります。 この状態では2つのリファレンスゲノムのどちらにもトラックが表示されていませんので、 「Open Track Selector」をクリックするか、赤い枠で囲ってあるメニューから 「View 1 track selector」もしくは「View 2 track selector」を選んでトラックを開いてください。 refSeqやToMMo Genome Variationなどのトラックを開くと、2つのリファレンスゲノムの比較が容易になります。
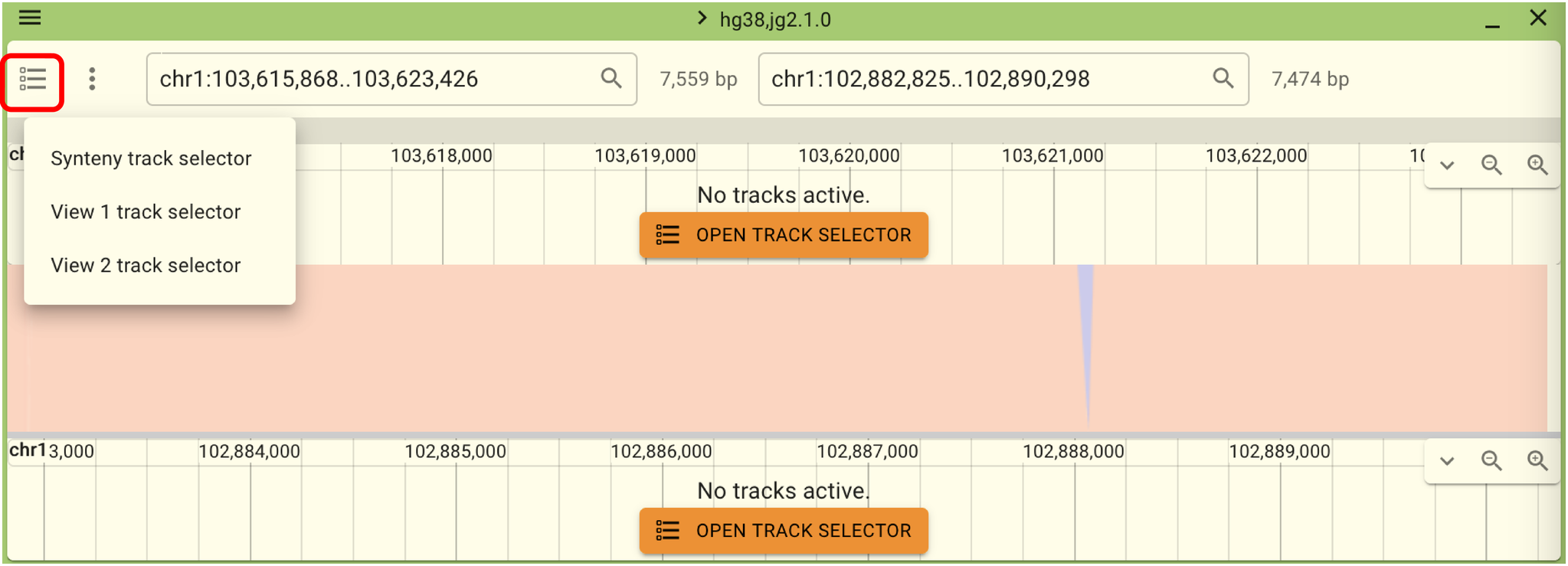
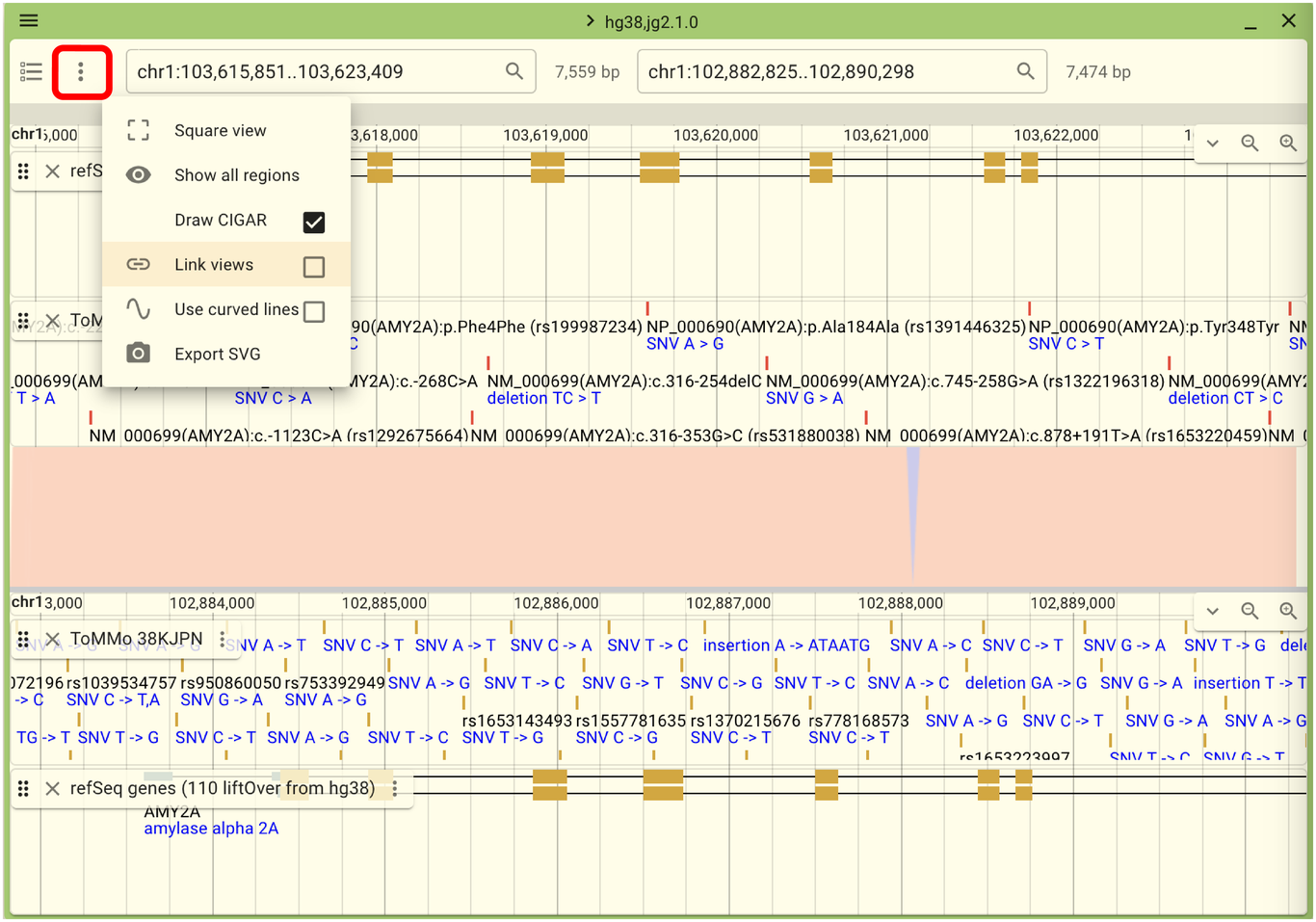
このままでは左右に動かしても上下のViewが連動しませんが、以下の画面の赤い枠のメニューをクリックし、 「Link views」を選択することで連動するようになります。
2.7.8. その他のJBrowse2の使い方
JBrowse 2の一般的な使い方については JBrowse2 user guide をご覧ください。