2.9. 代謝物相関ネットワークビューワー
jMorp には代謝物間の相関関係をネットワークとして可視化するツールが実装されています。 それが代謝物相関ネットワークビューワーです。この章ではこのツールの使い方を説明します。
2.9.1. 代謝物相関ネットワークビューワーの開き方
代謝物ページ の Correlation between other metabolites パネルから、 相関関係のネットワークビューワーを開くことができます。
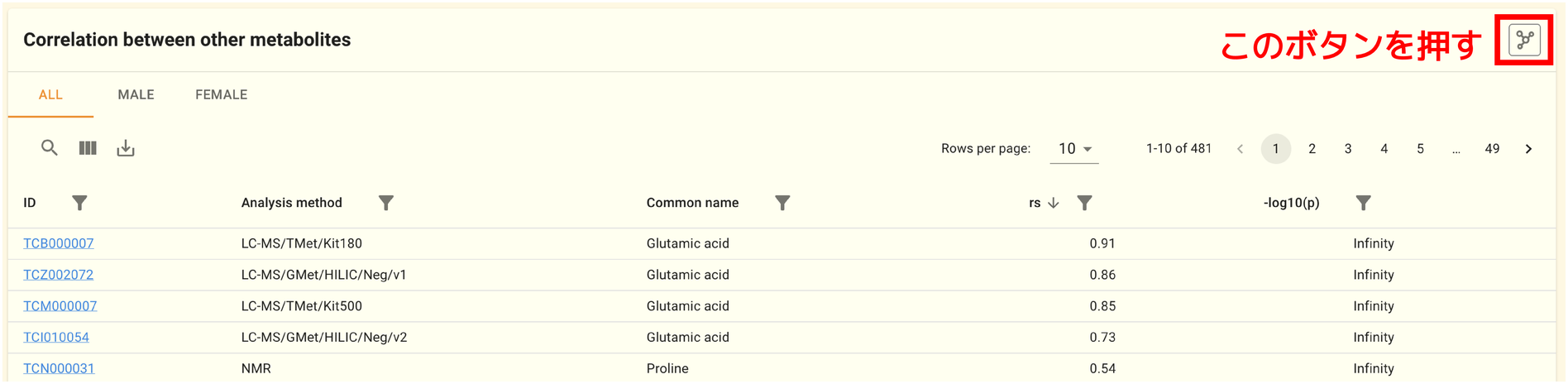
代謝物相関ネットワークビューワーは左側にネットワークを表示するためのパネル (ネットワークプロットパネル) があり、 右側にその表示をコントロールするためのパネル (ネットワーク情報パネル) や、 代謝物の詳細情報などを表示するためのパネル (選択対象詳細パネル) があります。
2.9.2. ネットワークプロットパネル
ネットワークをプロットしている左側にあるパネルです。代謝物間の相関関係をグラフィカルに確認できます。
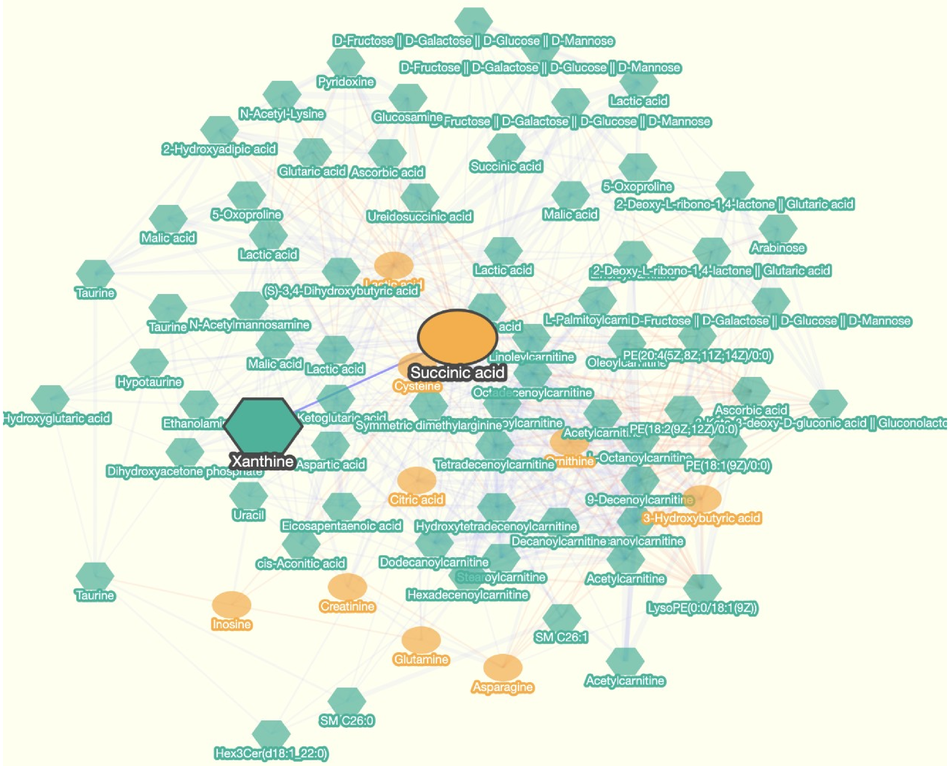
このパネルでは以下の操作ができます。
代謝物の選択: ノードをクリック
相関関係の選択: エッジをクリック(画像の例)
拡大縮小: スクロール(マウスホイールの回転)
代謝物の移動: ノードをドラッグ
全体の移動: ノード以外のところでドラッグ
注釈
プロットされるノードは "接続するエッジが1つ以上存在する ( 次数が1以上 )" という条件を満たす必要があります。 したがってエッジが一つも存在しないグラフは何も描画されません。
2.9.3. ネットワーク情報パネル
右上に表示されているパネルです。 VIEW ・ METABOLITES ・ EXPORT の3つのタブがあります。 ここでは相関ネットワーク全体に対するさまざまな操作を行えます。
2.9.3.1. ネットワークの操作
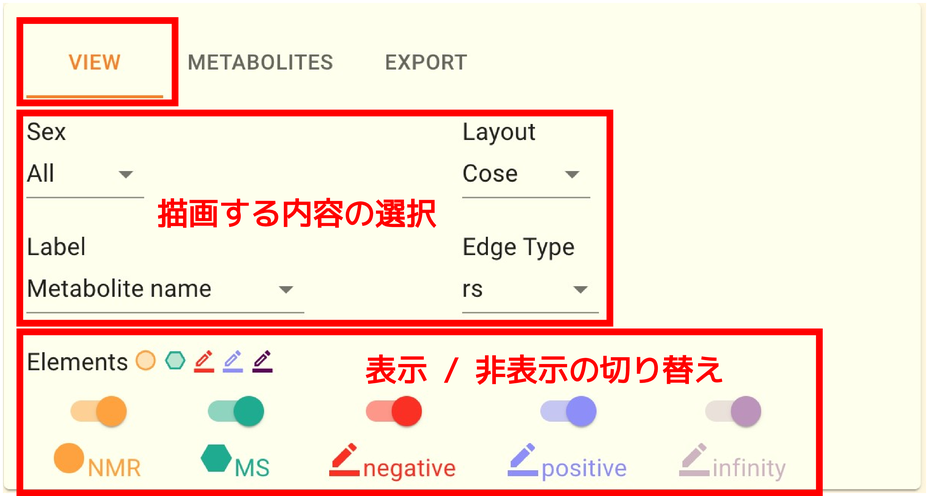
VIEW タブではネットワークとして可視化する情報を切り替えたり、ノードの配置を変更したりすることができます。 操作できるのは以下の5つです。
Sex: 性別条件をどうするか
Layout: 配置方法をどうするか
Label: 各ノードのラベルとして何を表示するか
Edge Type: ネットワークのエッジとして何の値を使うか
Elements: ネットワーク上のノードやエッジについて表示/非表示を切り替える
2.9.3.2. ネットワーク内の代謝物検索と表示切り替え
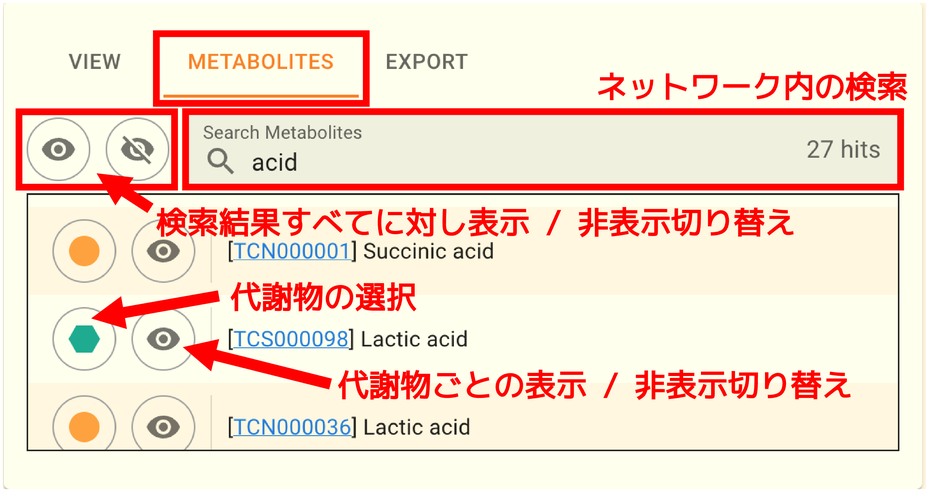
METABOLITES タブでは現在ネットワーク上に表示されている代謝物の一覧を見ることができます。 またネットワーク内の代謝物を検索および代謝物個別の表示・非表示を制御できます。 上記の画像は acid を検索した例です。検索にヒットした個数は検索欄の右端に表示されます。
2.9.3.3. ネットワークデータのダウンロード
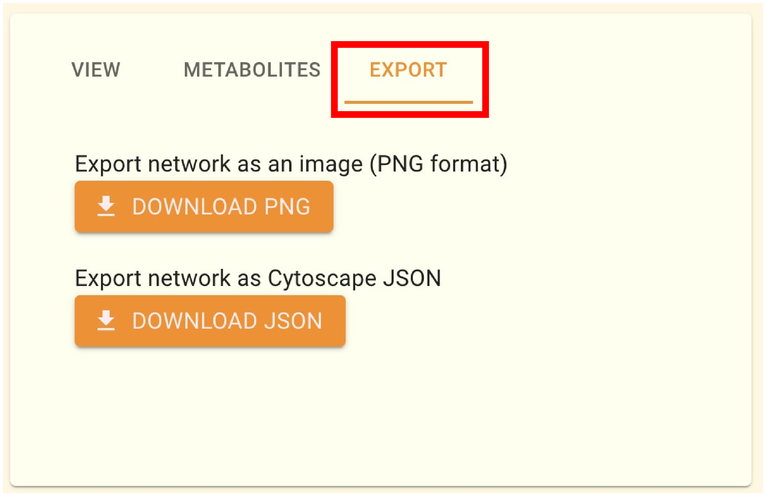
EXPORT タブでは、現在表示されている相関ネットワークの情報をダウンロードすることができます。 PNG 形式の画像データと、 JSON 形式のネットワーク構造データが取得可能です。 JSON 形式ファイルはデスクトップ版の Cytoscape で開くことができます。
2.9.4. 選択対象詳細パネル
現在ネットワーク上から選択しているもの (代謝物か相関関係か) のネットワークとしての詳細情報を右下のパネルから閲覧できます。 現在選択しているのが代謝物か相関関係かで表示される情報は異なります。
2.9.4.1. 代謝物の詳細
代謝物 (ノード) を選択している状態では以下のようなパネルになります。
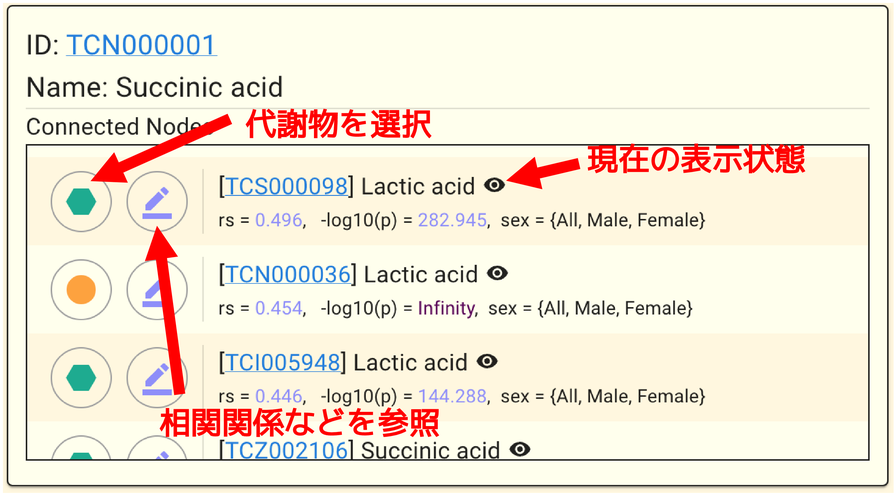
Connected Nodes では選択している代謝物と相関関係のデータがある代謝物一覧を確認できます。 リストは現在表示しているEdgeType ( rsか -log10(p)か ) の値で降順にソートされています。 リストの各項目左側には二つのボタンがあり、左はその代謝物を選択するボタン、右は相関関係を選択するボタンです。 またいくつかは背景が灰色になり右のボタンが押せないものがありますが、 それは現在表示している性別条件では相関関係のデータがないことを表しています。 代謝物名の右には現在ビューワー上に表示されているかどうかがわかるようになっており、 また代謝物名の下にはrsの値や-log10(p)の値、どの性別条件で相関関係のあるのかを示しています。
注釈
jMorp では Spearman 相関係数の絶対値が 0.2 以上かつ p 値が 10-5 以下の相関関係のみを掲載しています。 相関が無い は上記の条件を満たす相関関係が見つからないことを指します。
2.9.4.2. 相関関係の詳細
相関関係 (エッジ) を選択している状態では以下のようなパネルになります。
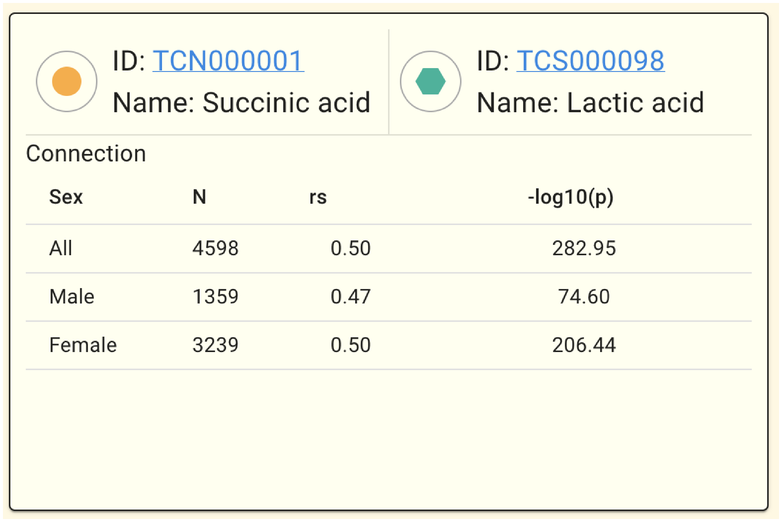
Connection では対象となる2つの代謝物の相関関係が各性別条件でどのようになっているかを確認できます。