2.3. SNV/INDEL アレル頻度比較ツール
SNV/INDEL アレル頻度比較ツールは、各民族集団ごとの SNV/INDEL アレル頻度比較をグラフィカルに行うものです。 対象の集団2個とゲノム上の領域を指定するとその領域に存在する SNV/INDEL を抽出し、そのアレル頻度を2軸でプロットします。
遺伝子ページ の SNV/INDELパネル などからアレル頻度比較ツールに移動することができます。 下の図は ALDH2上 の SNV/INDEL に対してアレル頻度比較ツールを実行 している様子です。
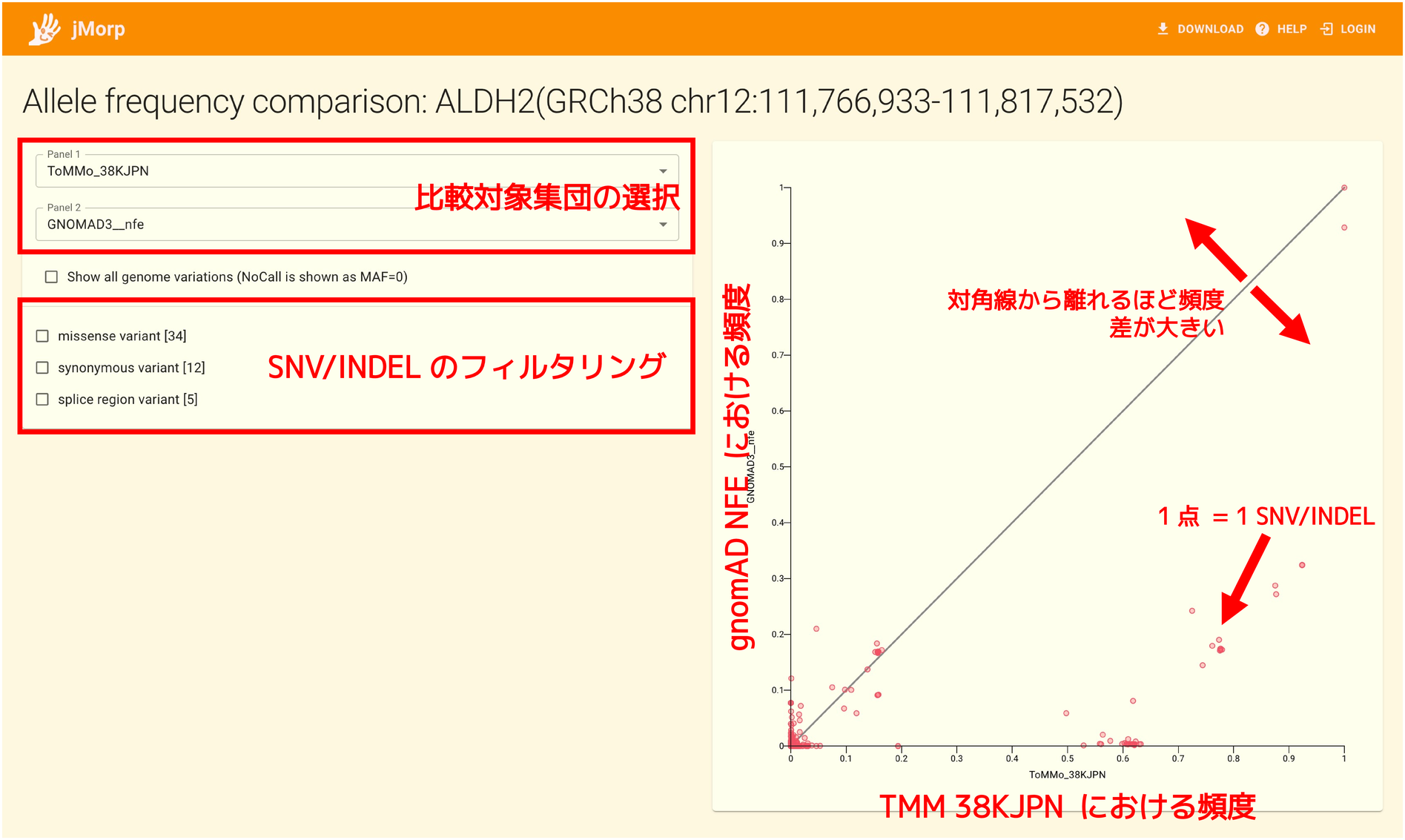
画面右上にあるセレクタで比較を行いたい2集団を選択します。 上図では ToMMo 38KJPN と gnomAD NFE (Non-Finnish European) が選択されています。 ここで選択された集団が画面右側のプロットの横軸と縦軸に対応します。
画面右側のプロットでは1点が1 SNV/INDEL に対応し、縦軸・横軸はそれぞれの集団における SNV/INDEL のアレル頻度を表します。
プロットには対角線が引かれています。プロット中の点が対角線に近い場合、2集団におけるSNV/INDELのアレル頻度は等しく、 逆に対角線から遠い場合は頻度差が大きいことを示します。 上の図では打たれている点の多くが対角線よりも下側に位置しているため、 ALDH2 上の SNV/INDEL のアレル頻度を ToMMo 38KJPN と gnomAD NFE で比べると ToMMo 38KJPN の方が高い傾向にある ということを見て取ることができます。
頻度差が大きい SNV/INDEL を具体的に知りたい場合は、マウスでプロット中の点をポイントすることで、 その点が対応するSNV/INDELや2集団におけるアレル頻度を確認することができます。 また画面左下に表示されている遺伝子アノテーションにチェックを入れることで、 選択されたアノテーションを持つ SNV/INDEL だけに絞ってプロットを行うことができます。