2.4. SNV/INDEL タンパク質構造マッピングツール
2.4.1. 基本的な使い方
jMorp にはゲノムバリアント (SNV/INDEL) がタンパク質構造のどこに当たるのかを示した タンパク質構造マッピングデータ が収録されています。 これは SNV/INDEL を DNA 配列上の位置から RNA 配列上の位置、アミノ酸配列上の位置に順次変換した後、 得られた配列を使って Protein Data Bank (PDB) や AlphaFold DB の アミノ酸配列データベースに対しBLAST検索を実行することでタンパク質上の位置を特定したものです。 タンパク質構造へのマッピングデータが存在する場合、遺伝子ページ や SNV/INDEL ページ から SNV/INDEL タンパク質構造マッピングツールへ移動し、SNV/INDEL と タンパク質構造の位置関係を見ることができます。
下の図は rs671 を PDB に収録されているタンパク質構造データにマッピングした結果を表示した時 の様子です。
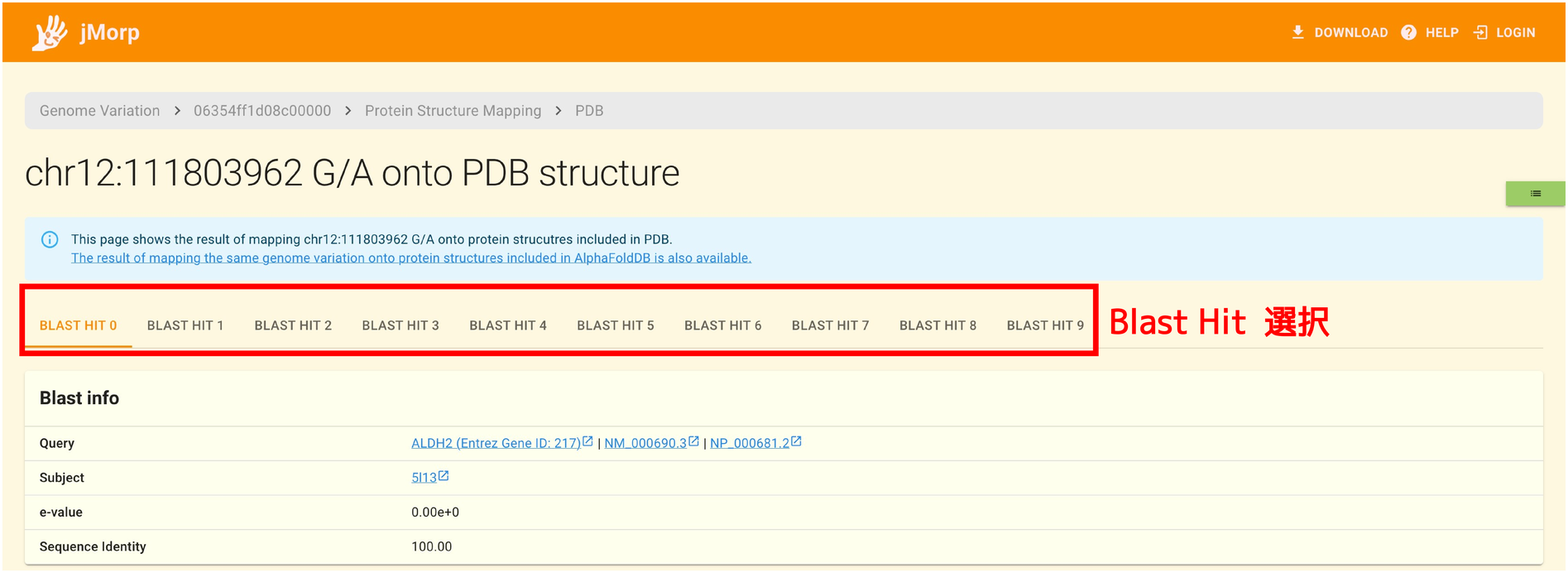
ページの最上部には BLAST HIT 0, BLAST HIT 1 ・・・と書かれたタブがあります。 前述の通り最終的には配列間の類似性を Blast で判定することで SNV/INDEL をタンパク質構造へマッピングしています。 この Blast のプロセスにおいては、1アミノ酸配列と複数のタンパク質構造がマッチされることがあります。 ページの最上部の BLAST HIT タブを用いることでマッチを切り替えることができます。
タブの下の Blast Info パネルでは前述した Blast 検索の結果 (クエリとして与えた配列、マッチしたタンパク質構造配列・ e-value ・ Sequence Identity など) が表示されます。
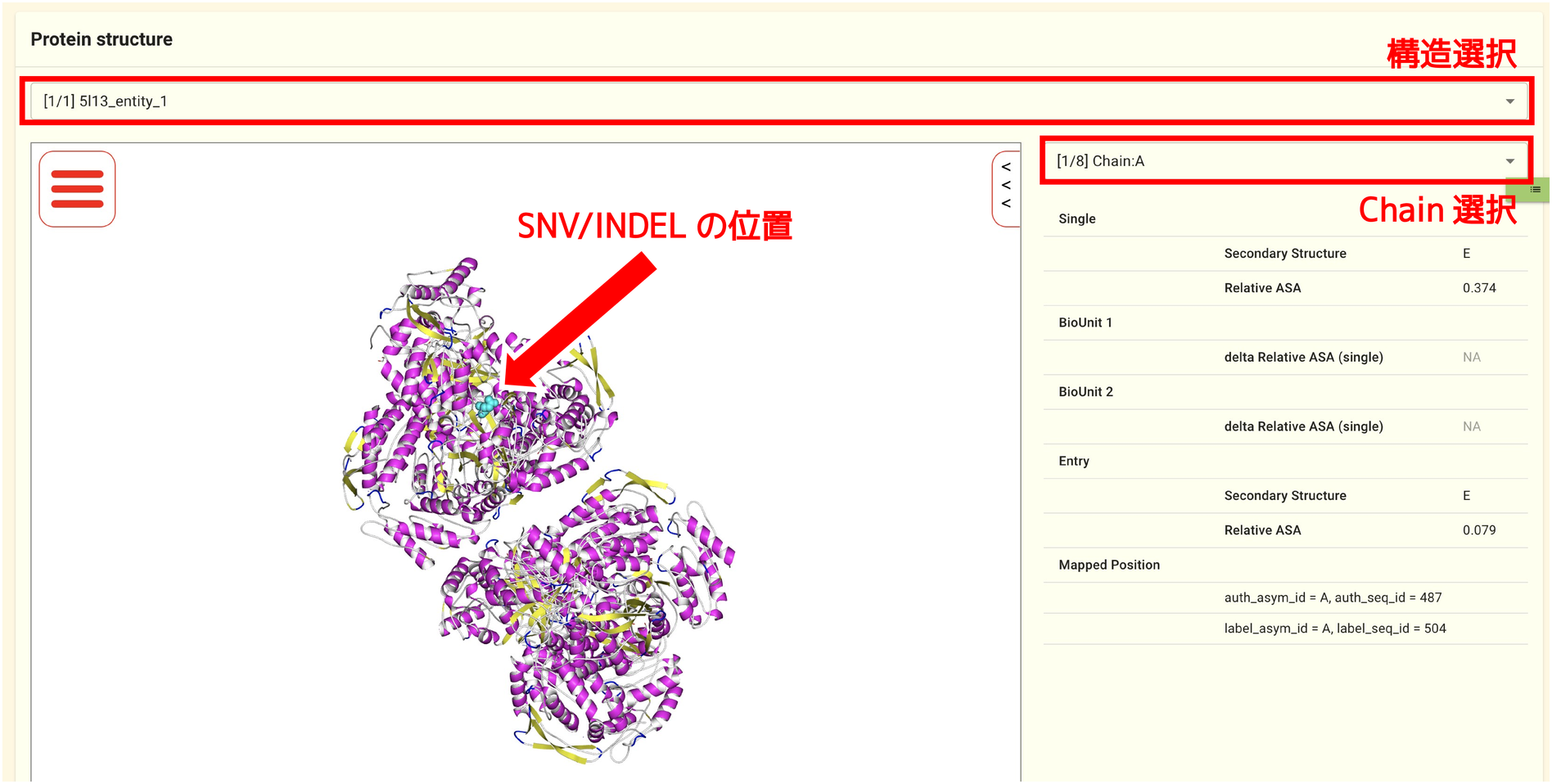
画面下部の Protein structure パネルではタンパク質構造モデルが表示され、どこに SNV/INDEL があるのかを見ることができます。 パネル上部のセレクタを使ってタンパク質構造を選択することができます。 jMorp のタンパク質構造ビューワーでは PDB に登録されているタンパク質構造をアミノ酸配列の類似性でグルーピングしており、 同じアミノ酸配列を持つタンパク質構造は1個の Blast Hit として現れます。 Protein structure の上部にあるセレクタを使うことでグルーピングされたタンパク質構造の中からどの構造に対して SNV/INDEL をマッピングした結果を表示するかを選択することができます。
Protein structure パネルの右側には Chain を選択するためのセレクタや構造の特性(二次構造や溶媒露出表面積 (Accessible Surface Area; ASA)) などが表示されます。
Protein structure パネルの左側には設定したタンパク質構造・ Chain における SNV/INDEL のマッピング結果が表示されます。 リボンモデルで表示されているタンパク質構造上に水色の球で SNV/INDEL の位置が表示されます。 タンパク質構造はマウス操作で回転・拡大・縮小することができます。
2.4.2. AlphaFold DB 収録タンパク質構造へのマッピング結果へ切り替え
jMorp には SNV/INDEL を PDB に収録されているタンパク質構造へマッピングした結果の他に、 SNV/INDEL を AlphaFold DB 収録のタンパク質へマッピングを行なった結果も収録されています。
データが存在する場合、ページ上部に次のような切り替えリンクが表示されますので このリンクを用いて AlphaFold DB 収録のタンパク質へマッピングを行なった結果へと切り替えることができます。
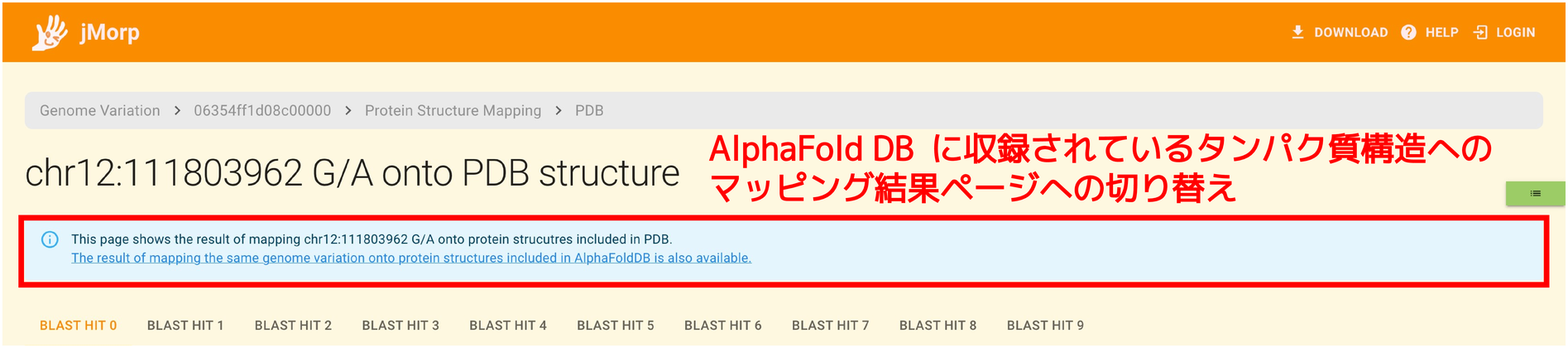
マッピング結果として表示される内容は PDB へのマッピング結果と同じ構成です。