2.1. 遺伝子ページ
jMorp では遺伝子ごとに、その遺伝子上にある SNV/INDEL ・メチル化サイト情報や遺伝子発現情報、GWAS解析結果情報など、 複数階層のデータを一括して閲覧できるページを用意しています。 トップページの検索ボックス から遺伝子シンボルや遺伝子 ID で検索を行うと、 この遺伝子ページが現れます。
遺伝子ページには以下のようなパネルが表示されます。
遺伝子情報パネル
SNV/INDEL パネル
SNV/INDEL 密度パネル
CNV パネル
メチル化情報パネル
遺伝子発現パネル
ゲノムブラウザパネル
GWAS解析結果パネル
HLAアレル頻度パネル (HLA遺伝子のみ)
PGxパネル (薬剤代謝関連遺伝子のみ)
ここから先の節では ALDH2 の遺伝子ページ (https://jmorp.megabank.tohoku.ac.jp/genes/217) を例にして パネルの内容を説明します。
2.1.1. 遺伝子情報パネル
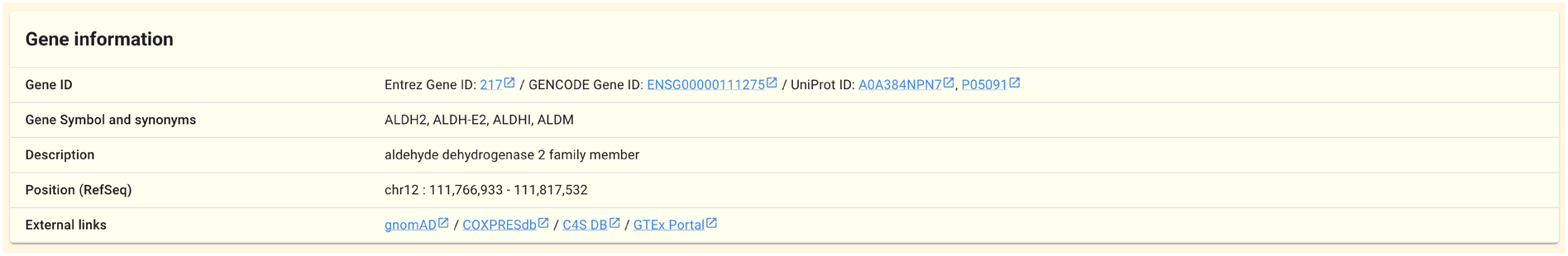
このパネルには遺伝子ID・シンボル等・ゲノム上の座標など、遺伝子に関する基礎的な情報が表示されます。 このパネルに表示される情報の多くは このページに記載のある公共データベース の情報を元にしています。
2.1.2. SNV/INDEL パネル
このパネルではTMMの短鎖全ゲノム解析によって検出されたSNV/INDEL のうち、この遺伝子上に存在するものを表示します。 1行が1SNV/INDELに対応します。
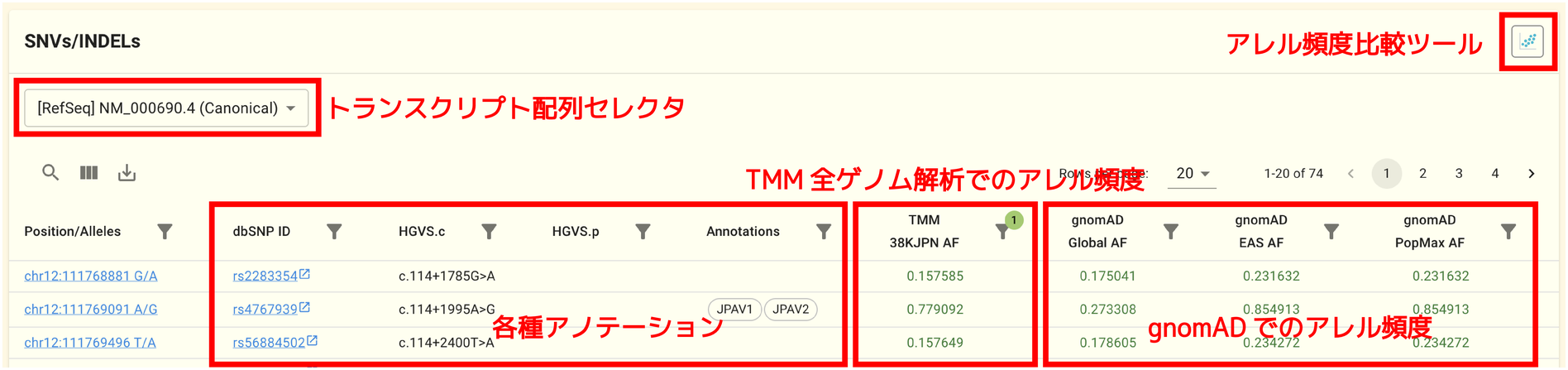
本書執筆時点では、アレル頻度情報として以下のデータが表示されます。
GRCh37: 8.3KJPN, 4.7KJPN, 3.5KJPNv2, gnomAD v2 Genome & Exome
GRCh38: 54KJPN, 38KJPN, 14KJPN, gnomAD v3 Genome
注釈
選択する参照ゲノムにより、SNV/INDEL パネルに表示されるアレル頻度データのソースが変わります。 参照ゲノムを切り替えるには、遺伝子ページの上部に表示されている以下のアラートの指示に従って下さい。

テーブル中の Position/Alleles などをクリックすることで SNV/INDEL ページ へ移動し、更に詳しい情報を確認することができます。
SNV/INDEL パネル右上のアイコンをクリックすると、アレル頻度比較ツールが起動します。 これは各集団ごとのアレル頻度をグラフィカルに比較できるツールです。 詳しい利用方法は こちらのページ で説明します。
このテーブルの上部にある屏風アイコン (四角が3個並んだアイコン) をクリックすることで バリアントの HGVS.c や HGVS.p などの追加情報を表示させることができます。 テーブルの操作方法の詳細については Tips: DataTable の操作方法 も合わせて参照してください。
テーブルには以下のような列が含まれています。
列名 |
内容 |
|---|---|
jMorp Variation ID |
jMorp 上で利用されている SNV/INDEL を識別するための ID |
jMorp Variation name |
jMorp 上で利用されている SNV/INDEL の名前 |
Position/Alleles |
参照ゲノム上での位置、および reference/alternative アレル |
dbSNP ID |
米 NCBI が運用する dbSNP データベースにおける ID |
HGVS.c |
cDNA 配列におけるバリアント表記 |
HGVS.p |
アミノ酸配列におけるバリアント表記 |
Annotations |
Gene annotation, ClinVar annotation, JPA を 1 セルに表示 |
Gene annotation |
遺伝子アノテーション |
ClinVar annotation |
ClinVar アノテーション |
JPA |
Japonica Array への搭載状況 |
TMM xxKJPN AF |
TMM の全ゲノムパネル (38KJPN など) におけるアレル頻度 |
TMM xxKJPN filter pass |
TMM の全ゲノムパネルににおいて、この SNV/INDEL が品質フィルタをパスしているかどうか |
gnomAD Global AF |
gnomAD におけるアレル頻度 |
gnomAD EAS AF |
gnomAD (East Asian 集団のみ) におけるアレル頻度 |
gnomAD PopMax AF |
gnomAD (PopMax; 民族集団ごとに頻度を算出し、その最大値を表示したもの) におけるアレル頻度 |
gnomAD filter pass |
gnomAD による全ゲノム解析において、この SNV/INDEL が品質フィルタをパスしているかどうか |
また、ゲノム上の変異をタンパク質構造上にマッピングした結果が利用可能な場合は、 HGVS.p に対してリンクが張られています。 このリンクをクリックすることでタンパク質構造ビューワーが開きます。タンパク質構造ビューワーに関する詳細は こちら を参照してください。
HGVS.c や HGVS.p は同じゲノムバリアントに対する物でもアノテーションに用いるトランスクリプト配列を変えると表記が変わります。 初期状態では RefSeq の Canonical transcript を用いてアノテーションした結果が表示されています。 テーブルの上部にあるセレクタを操作することでアノテーションに用いるトランスクリプト配列を変更することが可能です。
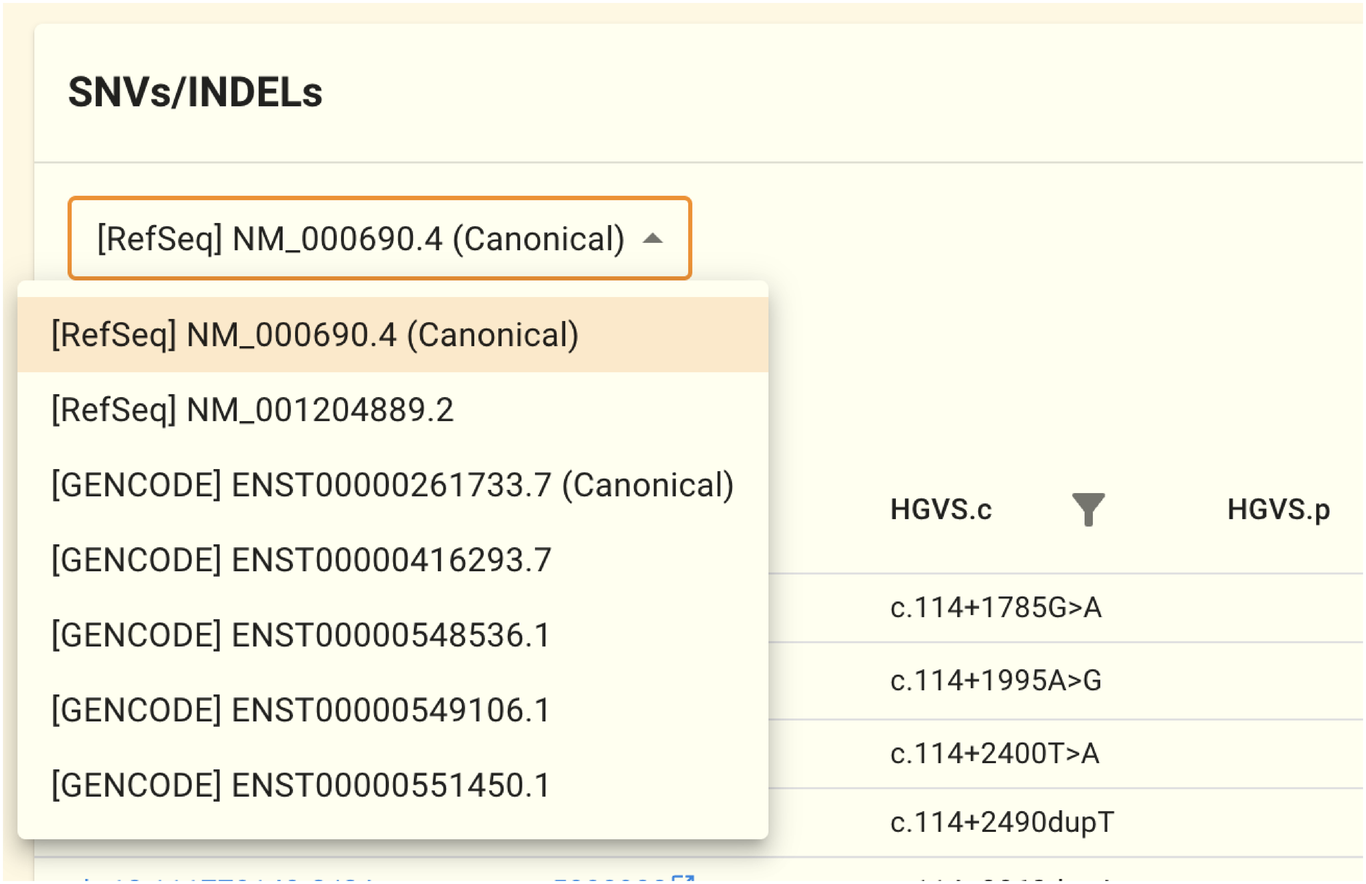
例えば ALDH2 であれば NM_000106.6 ・ NM_001025161.3 ・ ENST00000359033.4 ・ ENST00000360124.9 ・ ENST00000488442.1 ・ ENST00000645361.2 の合計6配列の登録があります。初期状態ではこの中から NM_000106.6 が選択されています。 このセレクタでの選択を変更することで、テーブル中の HGVS.c ・ HGVS.p 表記を選択したトランスクリプト配列のものに変更できます。
注釈
初期状態では RefSeq Canonical transcript を利用したアノテーションが自動的に表示されますが、 RefSeq よりも GENCODE の方が好みであれば、 初期表示を GENCODE の Canonical transcript へ変更することもできます。 詳しくは Preference ページ を参照して下さい。
警告
このセレクタによって HGVS.c 列・ HGVS.p 列の値は変更されますが、 jMorp Variation name 列の値は RefSeq Canonical transcript ベースのもののままです。 そのため、両者には齟齬がある可能性があります。
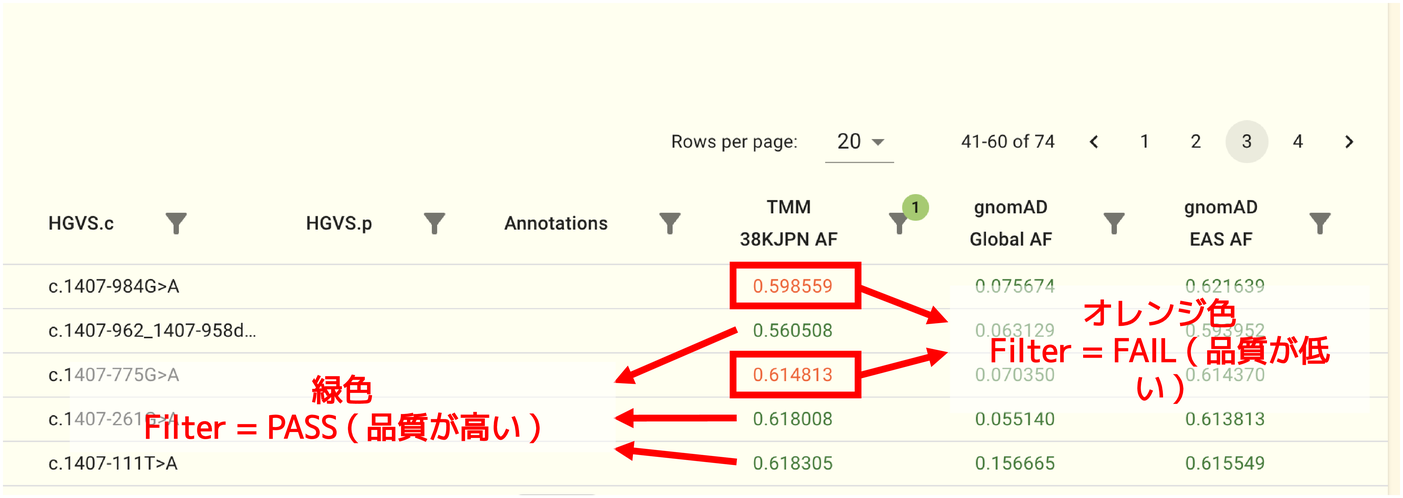
SNV/INDEL パネルに表示されるアレル頻度値には緑色で値が表示されるものとオレンジ色で値が表示されるものの2種類があります。 色の違いは SNV/INDEL の品質 (フィルタリング) に対応しています。緑色は品質が高いことを示し、オレンジ色は品質が低いことを示します。
注釈
TMM の全ゲノムパネルは、VQSR filtering の結果を反映しています。 フィルタリング手法の詳細は https://gatk.broadinstitute.org/hc/en-us/articles/360035531612 や DePristo et al. [13] を参照してください。 gnomAD データセットはTMMとは異なるフィルタリング手法が採用されています。詳細に関しては Karczewski et al. [14] を参照してください。
2.1.3. SNV/INDEL 密度パネル
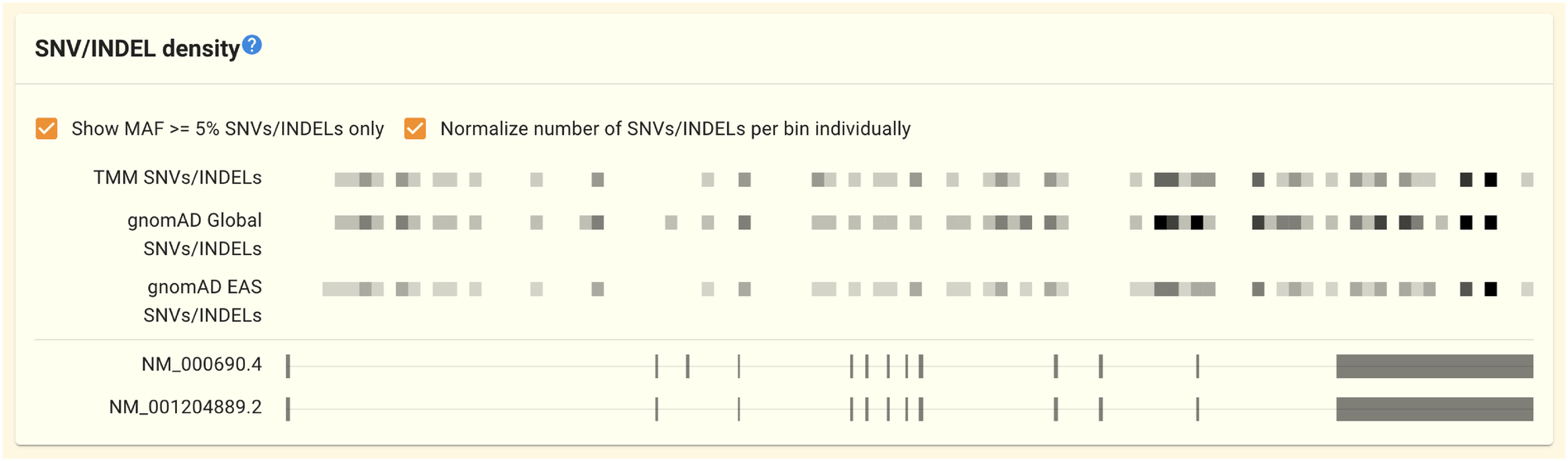
この節は鋭意執筆中です
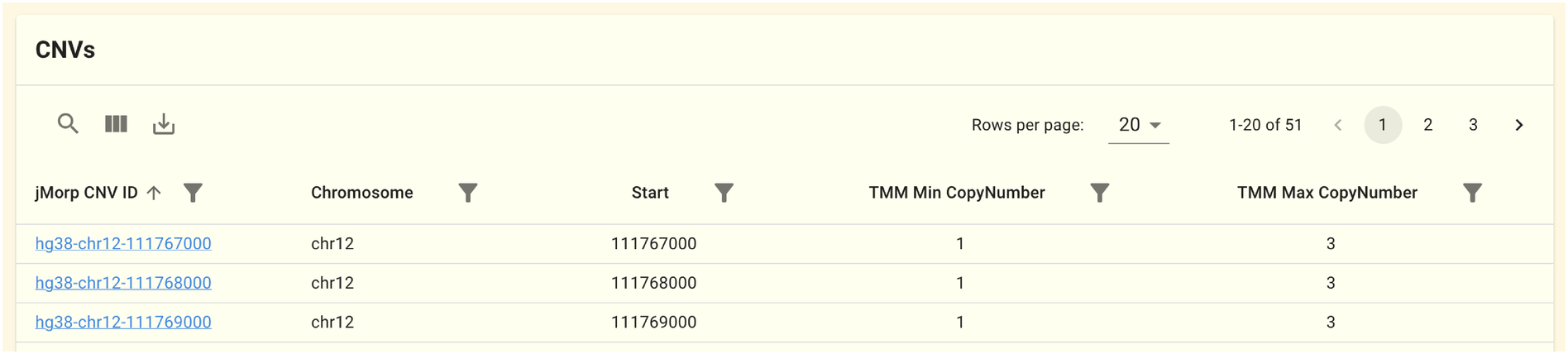
2.1.4. CNV パネル
このパネルにはこの遺伝子領域に重なるCNVの検出領域が表示されます。データは JCNVv1 データセットのものを元にしています。 ゲノム全体を1kbpごとにbinに切った領域単位でCNVを計算しており、このパネルには場所とコピー数の最小値、最大値が表示されています。 jMorp CNV IDをクリックすると、 CNVページ に移動し、コピー数のヒストグラムが表示されます。
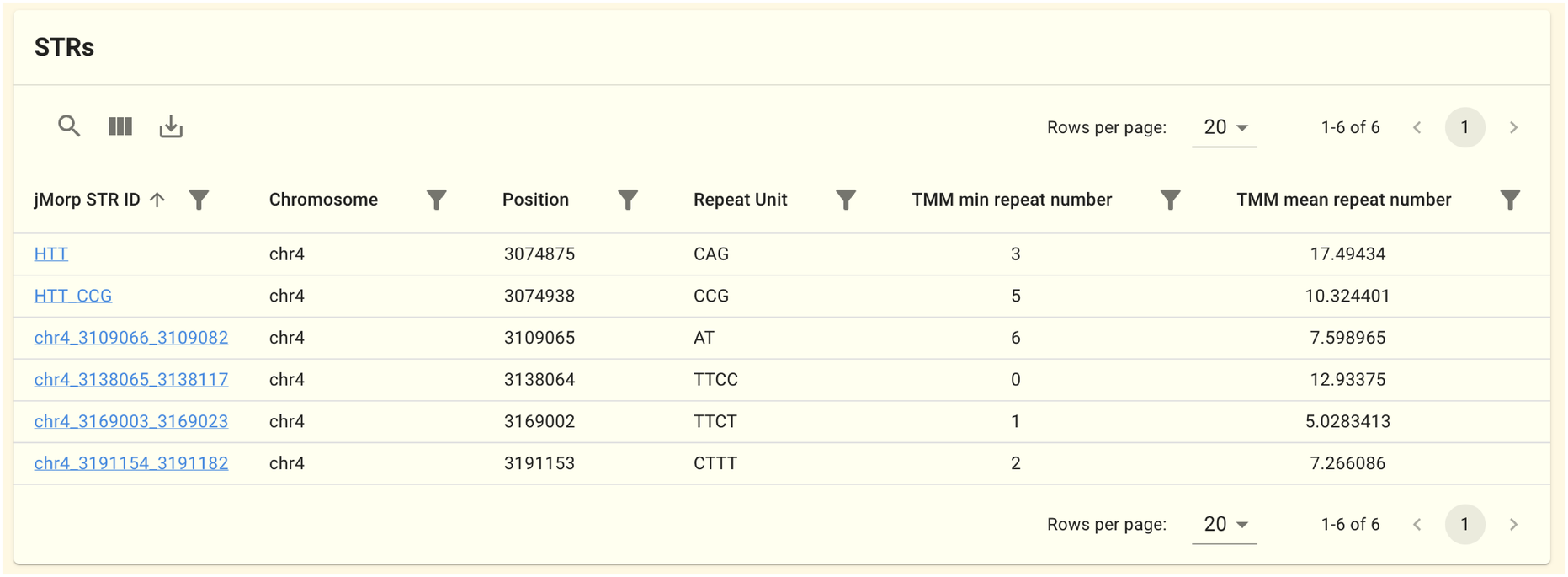
2.1.5. STR パネル
このパネルにはこの遺伝子領域に重なるShort Tandem Repeat (STR) の検出領域が表示されます。データは 54KJPN-STR データセットのものを元にしています。 STRは STR Catalogs に載っているものを利用しているため、遺伝子によっては重なっているSTRが存在しないために表示されない場合があります。 jMorp STR IDをクリックすると、 STRページ に移動し、リピート数のヒストグラムが表示されます。
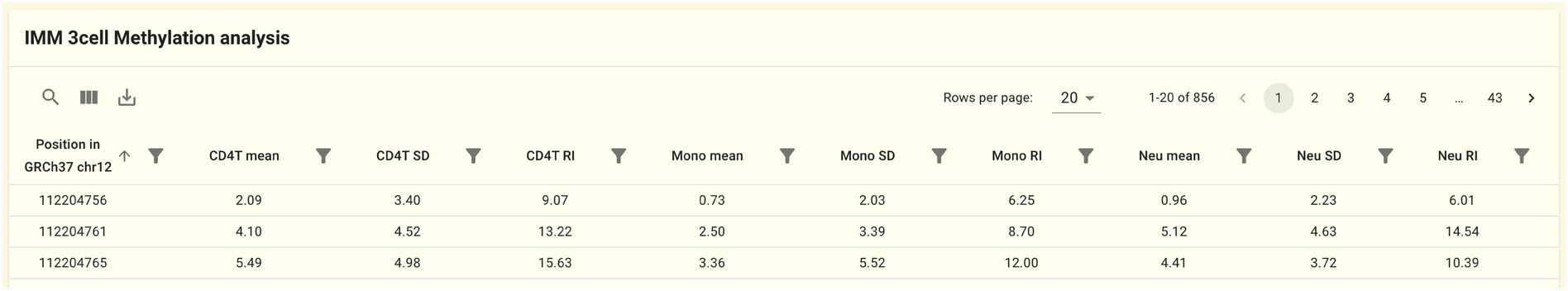
2.1.6. メチル化情報パネル
このパネルにはこの遺伝子上領域上で検出されたメチル化サイトの情報が表示されます。 データは IMM 3cell analysis データセットのものを元にしています。 ゲノムのポジションごと、かつ、細胞の種類 (CD4T・Mono・Neu) ごとにメチル化率の平均・分散・Reference Interval (RI) が表示されます。
注釈
IMM 3cell analysis データセットは GRCh37 を参照配列としてメチル化の解析を行ったものです。 そのため、GRCh37 版の遺伝子ページでのみこのパネルにデータが表示されます。 参照ゲノムを切り替えるには、遺伝子ページの上部に表示されている以下のアラートの指示に従って下さい。

2.1.7. 遺伝子発現パネル (ToMMo whole blood)
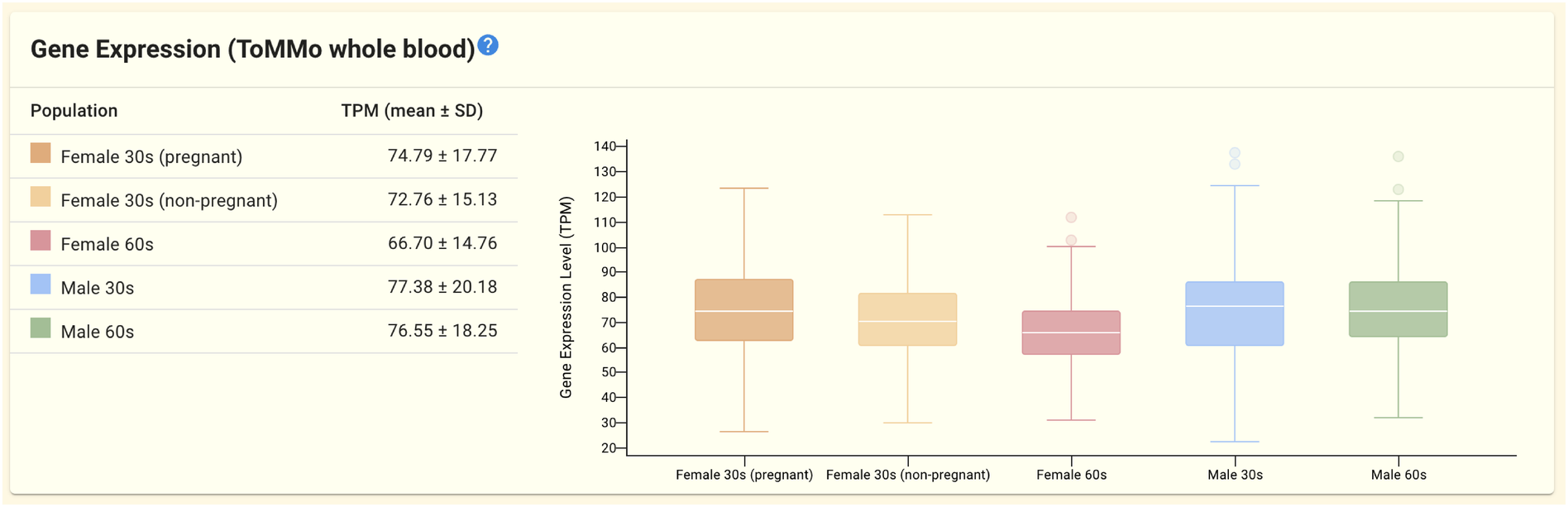
このパネルには ToMMo whole blood データセットに含まれる遺伝子発現データが表示されます。 性別および年齢で層別化した5つの集団 (30代妊娠女性, 30代非妊娠女性, 60代女性, 30代男性, 60代男性) について、 遺伝子発現量(TPM値)の統計量(平均、及び標準偏差)と箱ひげ図が表示されます。
2.1.8. 遺伝子発現パネル (IMM 3cell analysis)
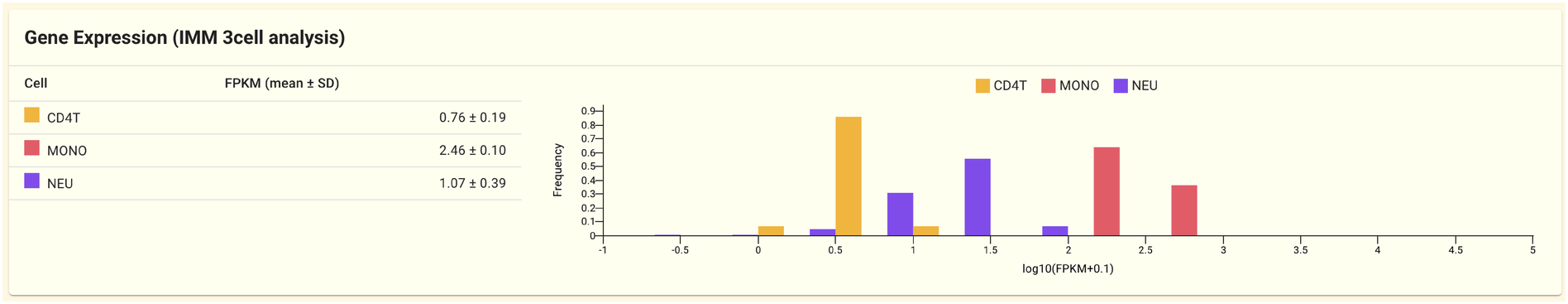
このパネルには IMM 3cell analyis データセットに含まれる遺伝子発現データが表示されます。 IMM 3cell analysis データセットは 細胞種 (CD4T・Mono・Neu) ごとに遺伝子発現を測定しています。
2.1.9. 遺伝子発現パネル (ToMMo ISO-Seq)

このパネルには ToMMo ISO-Seq データセットに含まれる遺伝子発現データが表示されます。 ToMMo ISO-Seq データセットは PacBio RS-II シークエンサによる 長鎖トランスクリプトーム解析 (Isoform sequencing; ISO-Seq) による遺伝子発現量データを含んでいます。
テーブルの1行が1トランスクリプトに対応し、細胞へのストレス有無別に FPKM値 (平均、及び標準偏差) が表示されます。
また、テーブルの右端にはそれぞれのトランスクリプトの構造を簡単に描いた図が表示されます。 四角がエクソンに対応し、マウスオーバーするとその座標が表示されます。
2.1.10. ゲノムブラウザパネル
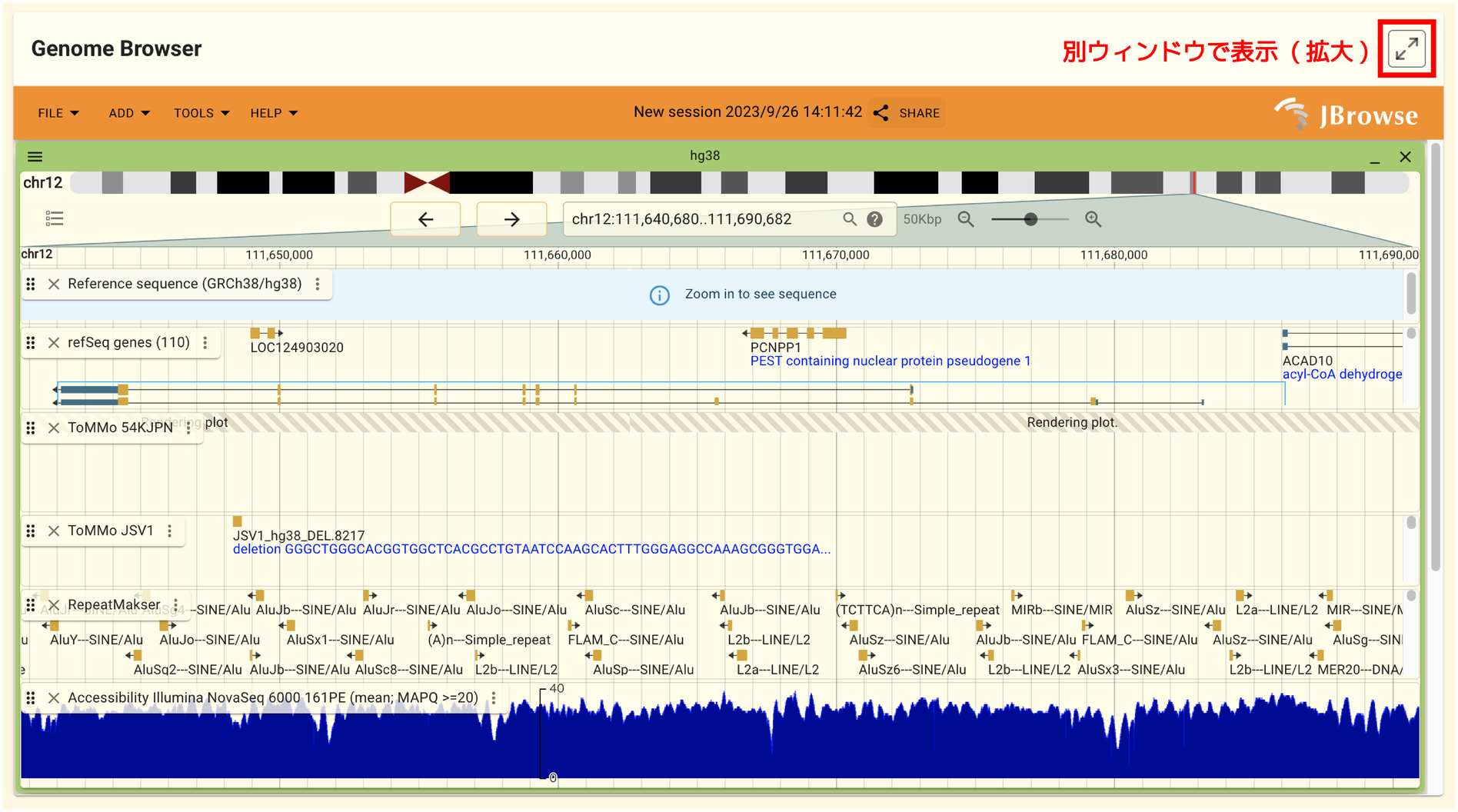
このパネルでは遺伝子領域をゲノムブラウザで閲覧することができます。 パネル右上の矢印アイコンをクリックすることでゲノムブラウザを別ウィンドウで表示することができます。 ゲノムブラウザの利用方法については 別の章 で解説します。
2.1.11. GWAS 解析結果パネル
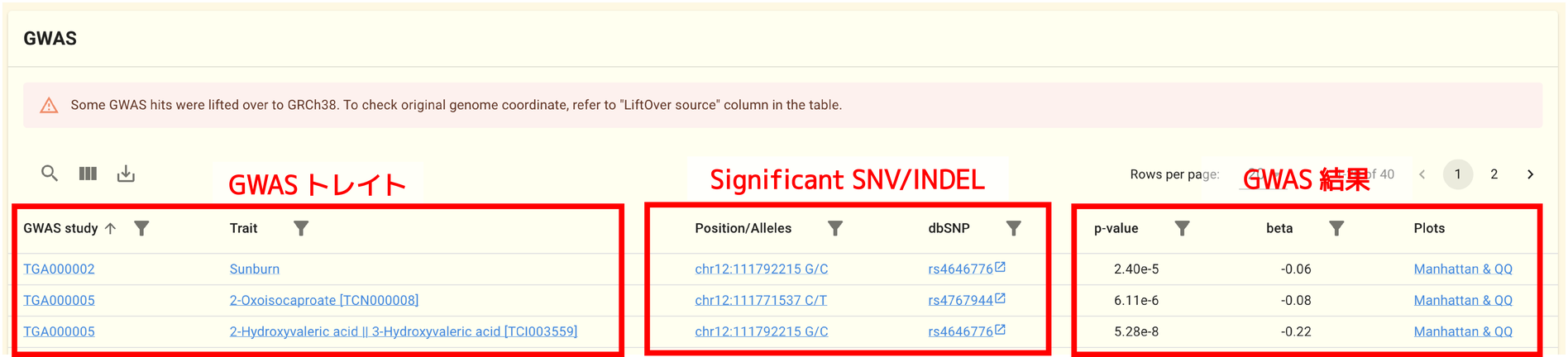
このパネルでは jMorp の GWAS 解析結果レポジトリからこの遺伝子に関係のある SNV/INDEL が抽出されて表示されます。
注釈
jMorp の GWAS 解析結果レポジトリに収録されている GWAS 要約統計量ファイルは GRCh37 の座標系で記述されているものが大半です。 遺伝子ページでは参照ゲノムの違いを意識すること無く GWAS 解析結果を検索・表示するため、 GWAS 解析結果パネルでは GWAS 要約統計量ファイル対して LiftOver が行われている場合があります。 この場合、テーブルの LiftOver source 列に元の GWAS 解析結果でのゲノム座標が表示されます。
2.1.12. HLA アレル頻度パネル
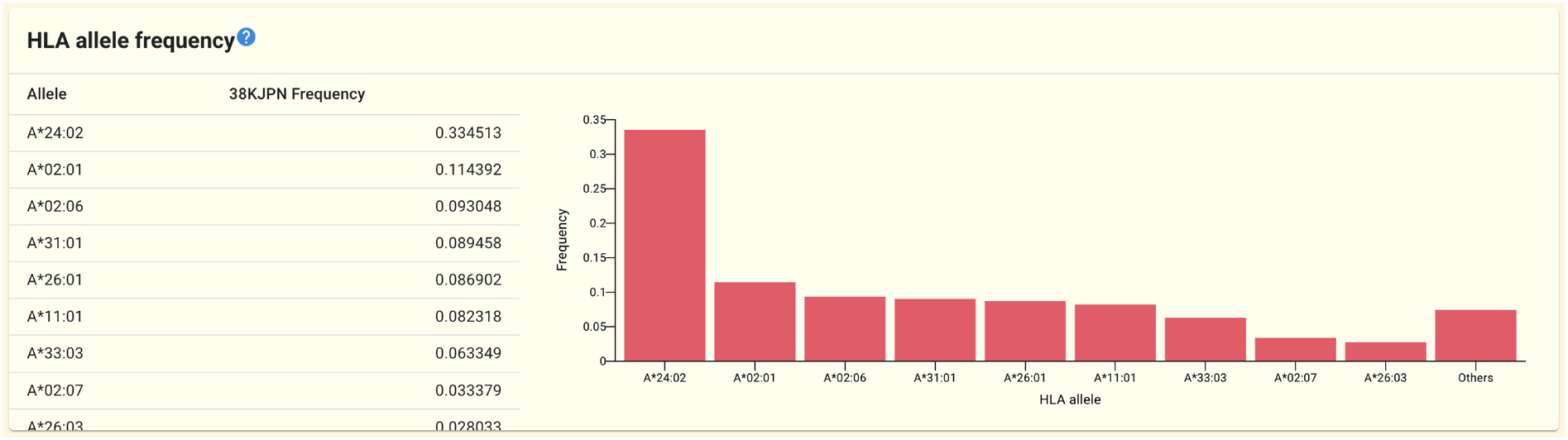
HLA-A や HLA-B など HLA 関連遺伝子に対応する遺伝子ページでは HLA アレル頻度パネルが表示されます。 54KJPN-HLA データセットのデータを元にしています。 上の図は HLA-A 遺伝子ページに表示される HLA アレル頻度パネルです。
2.1.13. PGx パネル
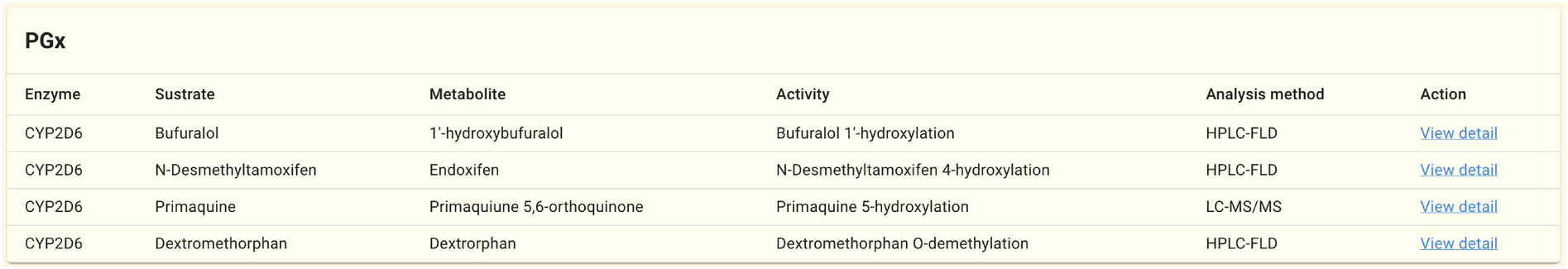
CYP2D6 など薬剤代謝関連遺伝子に対応する遺伝子ページでは PGx 解析結果へのリンクが表示されます。 PGx データの利用方法については PGx ページ 及び PGx データセット詳細 を参照して下さい。 上の図は CYP2D6 遺伝子ページに表示される PGx パネルです。